Startseite - Informatik aktuell · 2016. 2. 13. · Herausgeber Heinz Handels Universität zu...
Transcript of Startseite - Informatik aktuell · 2016. 2. 13. · Herausgeber Heinz Handels Universität zu...
-
Herausgegebenim Auftrag der Gesellschaft für Informatik (GI)
Weitere Bände in dieser Reihe:http://www.springer.com/series/2872
Informatik aktuell
-
Heinz Handels · Thomas M. DesernoHans-Peter Meinzer · Thomas TolxdorffHerausgeber
Bildverarbeitungfür die Medizin 2015
Algorithmen – Systeme – Anwendungen
Proceedings des Workshopsvom 15. bis 17. März 2015 in Lübeck
-
HerausgeberHeinz HandelsUniversität zu LübeckInstitut für Medizinische InformatikRatzeburger Allee 160, 23562 Lübeck
Thomas Martin Deserno, geb. LehmannUniklinik RWTH AachenInstitut für Medizinische InformatikPauwelsstr. 30, 52074 Aachen
Hans-Peter MeinzerDeutsches KrebsforschungszentrumAbteilung für Medizinische und Biologische Informatik Im Neuenheimer Feld 280, 69120 Heidelberg
Thomas TolxdorffCharité – Universitätsmedizin Berlin Institut für Medizinische Informatik Hindenburgdamm 30, 12200 Berlin
ISSN 1431-472XInformatik aktuellISBN 978-3-662-46223-2 ISBN 978-3-662-46224-9 (eBook) DOI 10.1007/978-3-662-46224-9
CR Subject Classification (1998): A.0, H.3, I.4, I.5, J.3, H.3.1, I.2.10, I.3.3, I.3.5, I.3.7, I.3.8, I.6.3
Die Deutsche Nationalbibliothek verzeichnet diese Publikation in der Deutschen Nationalbibliografie; detaillierte bibliografische Daten sind im Internet über http://dnb.d-nb.de abrufbar.
Springer Vieweg
© Springer-Verlag Berlin Heidelberg 2015
Das Werk einschließlich aller seiner Teile ist urheberrechtlich geschützt. Jede Verwertung, die nicht ausdrücklich vom Urheberrechtsgesetz zugelassen ist, bedarf der vorherigen Zustimmung des Verlags. Das gilt insbesondere für Vervielfältigungen, Bearbeitungen, Übersetzungen, Mikroverfilmungen und die Einspeicherung und Verarbeitung in elektronischen Systemen.
Die Wiedergabe von Gebrauchsnamen, Handelsnamen, Warenbezeichnungen usw. in diesem Werk berechtigt auch ohne besondere Kennzeichnung nicht zu der Annahme, dass solche Namen im Sinne der Warenzeichen- und Markenschutz-Gesetzgebung als frei zu betrachten wären und daher von jedermann benutzt werden dürften.
Der Verlag, die Autoren und die Herausgeber gehen davon aus, dass die Angaben und Informationen in diesem Werk zum Zeitpunkt der Veröffentlichung vollständig und korrekt sind. Weder der Verlag noch die Autoren oder die Herausgeber übernehmen, ausdrücklich oder implizit, Gewähr für den Inhalt des Werkes, etwaige Fehler oder Äußerungen.
Gedruckt auf säurefreiem und chlorfrei gebleichtem Papier
Springer-Verlag GmbH Berlin Heidelberg ist Teil der Fachverlagsgruppe Springer Science+Business Media (www.springer.com)
-
V
Bildverarbeitung für die Medizin 2015
Veranstalter
IMI Institut für Medizinische Informatik, Universität zu Lübeck
Unterstützende Fachgesellschaften
BVMI Berufsverband Medizinischer InformatikerCURAC Deutsche Gesellschaft für Computer- und Roboterassistierte
ChirurgieFachgruppe Medizinische Informatik der
DGBMT Deutschen Gesellschaft für Biomedizinische Technik imVDE Verband Deutscher ElektrotechnikerDAGM Deutsche Arbeitsgemeinschaft für MustererkennungGMDS Gesellschaft für Medizinische Informatik,
Biometrie und EpidemiologieIEEE Joint Chapter Engineering in Medicine and Biology,
German Section und derGI Gesellschaft für Informatik
Tagungsvorsitz
Prof. Dr. rer. nat. habil. Heinz HandelsInstitut für Medizinische Informatik, Universität zu Lübeck
Tagungssekretariat
Susanne PetersenInstitut für Medizinische Informatik, Universität zu LübeckPostanschrift: 23562 LübeckLieferanschrift: Ratzeburger Allee 160, Gebäude 64Telefon: +49 451 500 5601Telefax: +49 451 500 5610Email: [email protected]: http://bvm-workshop.org
Lokales BVM-Komitee
Prof. Dr. H. Handels (Leitung)Prof. Dr. J. Barkhausen, Klinik für Radiologie und NuklearmedizinProf. Dr. T. Buzug, Institut für MedizintechnikProf. Dr. J. Modersitzki, Fraunhofer MEVIS Lübeck
-
VI
Verteilte BVM-Organisation
Prof. Dr. Thomas M. Deserno, Jan DovermannRheinisch-Westfälische Technische Hochschule Aachen (Tagungsband)
Prof. Dr. Heinz Handels, Dr. Jan-Hinrich WrageUniversität zu Lübeck (Beitragsbegutachtung)
Prof. Dr. Hans-Peter Meinzer, Michael BrehlerDeutsches Krebsforschungszentrum Heidelberg (Anmeldung)
Prof. Dr. Thomas Tolxdorff, Dr. Thorsten SchaafCharité – Universitätsmedizin Berlin (Internetpräsenz)
Programmkomitee
Prof. Dr. Dr. Johannes Bernarding, Universität MagdeburgProf. Dr. Oliver Burgert, Universität LeipzigProf. Dr. Thorsten Buzug, Universität zu LübeckProf. Dr. Thomas Deserno, RWTH AachenProf. Dr. Hartmut Dickhaus, Universität HeidelbergDr. Jan Ehrhardt, Universität zu LübeckProf. Dr. Nils Forkert, University of Calgary, KanadaProf. Dr. Rolf-Rainer Grigat, TU Hamburg-HarburgProf. Dr. Heinz Handels, Universität zu LübeckPriv.-Doz. Dr. Peter Hastreiter, Universität ErlangenJun.-Prof. Dr. Mattias Heinrich, Universität zu LübeckDr. Tobias Heimann, Siemens Corporate Technology ErlangenProf. Dr. Joachim Hornegger, Universität ErlangenProf. Ron Kikinis, MD, Fraunhofer MEVIS BremenDr. Cristian Lorenz, PHILIPS HamburgDr. Klaus Maier-Hein, DKFZ HeidelbergPriv.-Doz. Dr. Lena Maier-Hein, DKFZ HeidelbergProf. Dr. Hans-Peter Meinzer, DKFZ HeidelbergProf. Dr. Dorit Merhof, RWTH AachenProf. Dr. Alfred Mertins, Universität zu LübeckProf. Dr. Jan Modersitzki, Fraunhofer MEVIS LübeckProf. Dr. Heinrich Müller, Technische Unversität DortmundProf. Dr. Henning Müller, Université Sierre SchweizProf. Dr. Nassir Navab, Technische Universität MünchenProf. Dr. Arya Nabavi, International Neuroscience Institute, HannoverProf. Dr. Heinrich Niemann, Universität ErlangenProf. Dr. Christoph Palm, OTH RegensburgProf. Dr. Bernhard Preim, Universität MagdeburgPriv.-Doz. Dr. Karl Rohr, Universität HeidelbergPriv.-Doz. Dr. Dennis Säring, Universitätsklinikum HamburgProf. Ingrid Scholl, Fachhochschule AachenDr. Stefanie Speidel, KIT Karlsruhe
-
VII
Prof. Dr. Thomas Tolxdorff, Charité-Universitätsmedizin BerlinDr. Gudrun Wagenknecht, Forschungszentrum JülichDr. Stefan Wesarg, Fraunhofer IGD DarmstadtProf. Dr. Herbert Witte, Universität JenaPriv.-Doz. Dr. Thomas Wittenberg, Fraunhofer IIS ErlangenDr. Stefan Wörz, Universität HeidelbergProf. Dr. Ivo Wolf, Hochschule Mannheim
-
VIII
Sponsoren des Workshops BVM 2015
Die BVM wäre ohne die finanzielle Unterstützung der Industrie in ihrer so er-folgreichen Konzeption nicht durchführbar. Deshalb freuen wir uns sehr überlangjährige kontinuierliche Unterstützung mancher Firmen sowie auch über dasneue Engagement anderer. Dies gilt insbesondere für unsere Platinsponsoren.
Platin-Sponsor
Agfa HealthCareKonrad-Zuse-Platz 1-3, D-53227 Bonnhttp://www.agfa.com/healthcare
OLYMPUS SURGICAL TECHNOLOGIES EUROPEOlympus Winter und Ibe GmbHKuehnstrasse 61, D-22045 Hamburghttp://www.olympus-oste.eu
Sponsoren
CHILI GmbHBurgstr. 61, D-69121 Heidelberghttp://www.chili-radiology.com
Haption GmbHTechnologiezentrum am Europaplatz Dennewartstr. 25, D-52068 Aachenhttp://www.haption.de
MiE GmbHHauptstrasse 112, D-23845 Sethhttp://www.miegermany.de
Springer Verlag GmbHTiergartenstr. 17, D-69121 Heidelberghttp://www.springer.com
VISUS Technology Transfer GmbHUniversitätsstr. 136, D-44799 Bochumhttp://www.visus.com
-
IX
Preisträger des BVM-Workshops 2014 in Aachen
Auf der BVM 2014 wurde der mit 1.000 e dotierte BVM-Award an eine heraus-ragende Diplom-, Bachelor-, Master- oder Doktorarbeit aus dem Bereich der Me-dizinischen Bildverarbeitung gesplittet. Die mit einem Preisgeld von je 333,33 edotierten BVM-Preise zeichnen besonders hervorragende Arbeiten aus, die aufdem Workshop präsentiert wurden.
BVM-Award 2014 für eine herausragende Dissertation
Dagmar Kainmüller (Universität zu Lübeck)Deformable Meshes for Accurate Automatic Segmentation of Medical Image Da-taNils D. Forkert (Universität Hamburg)Model-Based Analysis of Cerebrovascular Diseases Combining 3D and 4D MRADatasets
BVM-Preis 2014 für die beste wissenschaftliche Arbeit
Thomas Köhler (Friedrich-Alexander University Erlangen-Nürnberg)Outlier Detection for Multi-Sensor Super-Resolution in Hybrid 3D Endoscopy
BVM-Preis 2014 für den besten Vortrag
Sandy Engelhardt mit Bastian Graser, Raffaele De Simone, Norbert Zimmer-mann, Matthias Krack, Hans-Peter Meinzer, Diana Nabers, Ivo Wolf (DKFZHeidelberg)Vermessung des Mitralapparats mit einem optisch getrackten Zeigeinstrumentfür die virtuelle Annuloplastie
BVM-Preis 2014 für die beste Posterpräsentation
Julian Schröder mit Andre Mastmeyer, Dirk Fortmeier, Heinz Handels (Univer-sität zu Lübeck)Ultraschallsimulation für das Training von Gallengangspunktionen
-
XI
Vorwort
In diesem Jahr wird die Tagung Bildverarbeitung für die Medizin (BVM 2015)vom Institut für Medizinische Informatik an der Universität zu Lübeck ausge-richtet. Nach der erfolgreichen Durchführung der BVM 2001 und 2011 findetdiese zentrale Tagung zu neuen Entwicklungen in der Medizinischen Bildverar-beitung in Deutschland nun zum dritten Mal in der traditionsreichen HansestadtLübeck statt.
Die Bedeutung des Themas Bildverarbeitung für die Medizin hat über dieJahre deutlich zugenommen. Die Bildverarbeitung ist eine Schlüsseltechnologiein verschiedenen medizinischen Bereichen wie der Diagnoseunterstützung, derOP-Planung und der bildgeführten Chirurgie. An der Universität zu Lübeckbilden die Medizinische Bildgebung und Bildverarbeitung einen zentralen For-schungsschwerpunkt, der in den letzten Jahren systematisch ausgebaut wurde.Vor diesem Hintergrund ist es eine besondere Freude, die BVM 2015 in Lübeckausrichten zu dürfen.
Die BVM konnte sich in den letzten Jahren als ein zentrales interdiszipli-näres Forum für die Präsentation und Diskussion von Methoden, Systemen undAnwendungen im Bereich der Medizinischen Bildverarbeitung etablieren. Zielder Tagung ist die Darstellung aktueller Forschungsergebnisse und die Vertie-fung der Gespräche zwischen Wissenschaftlern, Industrie und Anwendern. DieBVM richtet sich ausdrücklich auch an Nachwuchswissenschaftler, die über ihreBachelor-, Master-, Promotions- und Habilitationsprojekte berichten wollen.
Die BVM 2015 wird unter der Federführung von Prof. Dr. rer. nat. habil.Heinz Handels, Direktor des Instituts für Medizinische Informatik der Univer-sität zu Lübeck, ausgerichtet. Die Organisation ist wie in den letzten Jahrenauf Fachkollegen aus Aachen, Berlin, Heidelberg und Lübeck verteilt, so dassdie Organisatoren der vergangenen Jahre ihre Erfahrungen mit einfließen lassenkönnen.
Anhand anonymisierter Bewertungen durch jeweils drei Fachgutachter wur-den 86 Beiträge zur Präsentation ausgewählt: 48 Vorträge, 34 Poster und 4 Soft-waredemonstrationen. Die Qualität der eingereichten Arbeiten war insgesamtsehr hoch. Die besten Arbeiten werden auch im Jahr 2015 mit BVM-Preisenausgezeichnet. Die schriftlichen Langfassungen der Beiträge werden im Tagungs-band abgedruckt, der auch dieses Jahr wieder im Springer Verlag in der ReiheInformatik aktuell zur BVM erscheint.
Höhepunkte der BVM 2015 bilden die Gastvorträge von Prof. Dr. JuliaSchnabel, Institute of Biomedical Engineering der University of Oxford (UK)und von Prof. Dr. Thorsten Buzug, Institut für Medizintechnik der Universitätzu Lübeck, die neueste Methoden und Trends in der medizinischen Bildregistrie-rung und Modellierung sowie aktuelle und zukünftige Entwicklungen der neuenBildgebungstechnik Magnetic Particle Imaging vorstellen.
Die Internetseiten des Workshops bieten ausführliche Informationen über dasProgramm und organisatorische Details rund um die BVM 2015. Sie sind abruf-bar unter der Adresse:
-
XII
http://www.bvm-workshop.org
Am Tag vor dem wissenschaftlichen Programm werden zwei Tutorials ange-boten:Prof. Dr. Bernhard Preim, Institut für Simulation und Graphik der Otto-von-Guericke-Universität Magdeburg, hält unterstützt von seinen Mitarbeiterinnenund Mitarbeitern ein Tutorial zum Thema
”Visualisierung und Virtual-Reality-
Techniken in der Medizin“. Neben grundlegenden Methoden der medizinischenVisualisierung wie dem Volume Rendering und der Oberflächenvisualisierungwerden ihre Anwendungsmöglichkeiten in verschiedenen medizinischen Berei-chen vorgestellt. Weiterhin werden ausgewählte Anwendungsbeispiele aus derOperations- und Interventionsplanung sowie Methoden zur intraoperativen Vi-sualisierung mit Virtual-Reality-Techniken präsentiert.
Das zweite Tutorial trägt den Titel”Medizinische Bildregistrierung“. Prof.
Dr. Jan Modersitzki, Fraunhofer MEVIS Lübeck, erläutert hier unterstützt vonseinen Mitarbeiterinnen und Mitarbeitern wichtige medizinische Bildregistrie-rungsmethoden, beleuchtet ihren mathematischen Hintergrund und zeigt anhandvon Beispielen ihre vielfältigen medizinischen Anwendungsmöglichkeiten auf.
Die Herausgeber dieser Proceedings möchten allen herzlich danken, die zumGelingen der BVM 2015 beigetragen haben. Den Autoren für die rechtzeiti-ge und formgerechte Einreichung ihrer qualitativ hochwertigen Arbeiten, demProgrammkomitee für die gründliche Begutachtung, den Gastrednern und denReferenten der Tutorials für Ihre aktive Mitgestaltung und inhaltliche Bereiche-rung der BVM 2015. Herrn Dr. Thorsten Schaaf vom Institut für MedizinischeInformatik der Charité Universitätsmedizin Berlin danken wir für die engagierteMithilfe bei der Erstellung und Pflege der Internetpräsentation. Herrn MichaelBrehler von der Abteilung Medizinische und Biologische Informatik am Deut-schen Krebsforschungszentrum in Heidelberg möchten wir herzlich für seine en-gagierte Tätigkeit bei der Umsetzung der WWW-basierten Tagungsanmeldungund der Pflege des BVM-Email-Verteilers danken. Herrn Jan Dovermann vomInstitut für Medizinische Informatik der RWTH Aachen danken wir für die tat-kräftige Mitarbeit bei der Erstellung des Proceedingsbandes. Für die webbasier-te Durchführung des Reviewingprozesses gebührt Herrn Dr. Jan-Hinrich Wrage,für die Programmerstellung Herrn Dr. Jan Ehrhardt und Jun.-Prof. Dr. Matti-as Heinrich vom Institut für Medizinische Informatik der Universität zu Lübeckunser Dank. Weiterhin danken wir der Tagungssekretärin Frau Susanne Petersenund allen übrigen Mitarbeiterinnen und Mitarbeitern des Instituts für Medizi-nische Informatik der Universität zu Lübeck für ihre tatkräftige Unterstützungbei der Organisation und Durchführung der BVM 2015. Unser Dank gilt auchden Mitgliedern des lokalen Lübecker BVM-Komitees für die Unterstützung beiWerbemaßnahmen und der Gewinn von Industriesponsoren für die BVM 2015.Für die finanzielle Unterstützung bedanken wir uns bei den Fachgesellschaftenund der Industrie.
Wir wünschen allen Teilnehmerinnen und Teilnehmern der BVM 2015 lehr-reiche Tutorials, viele anregende Vorträge, Gespräche an den Postern und in
-
XIII
der Industrieausstellung sowie interessante neue Kontakte zu Kolleginnen undKollegen aus dem Bereich der Medizinischen Bildverarbeitung.
Januar 2015 Heinz Handels (Lübeck)Thomas Deserno (Aachen)
Hans-Peter Meinzer (Heidelberg)Thomas Tolxdorff (Berlin)
-
Inhaltsverzeichnis
Die fortlaufende Nummer am linken Seitenrand entspricht den Beitragsnum-mern, wie sie im endgültigen Programm des Workshops zu finden sind. Dabeisteht V für Vortrag, P für Poster und S für Softwaredemonstration.
Eingeladene Vorträge
V1 Schnabel J: Complex Motion Modelling in Cancer Imaging . . . . . . . . . 1
V2 Buzug TM: Magnetic Particle Imaging . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 3
Segmentierung I
V3 Kottke D, Gulamhussene G, Tönnies K: Data-Driven SpineDetection for Multi-Sequence MRI . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 5
V4 Ivanovska T, Wang L, Völzke H, Hegenscheid K: Automated BreastVolume of Interest Selection by Analysing Breast-Air Boundaries inMRI . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 11
V5 Kleinfeld A, Maier O, Forkert N, Handels H: AutomatischeDetektion von Okklusionen zerebraler Arterien in3D-Magnetresonanzangiographiedaten . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 17
V6 Hoffmann M, Müller S, Kurzidim K, Strobel N, Hornegger J:Robust Identification of Contrasted Frames in Fluoroscopic Images 23
V7 Lüddemann T, Egger J: Interaktive und skalierungsinvarianteSegmentierung des Rektums/Sigmoid in intraoperativenMRT-Aufnahmen für die gynäkologische Brachytherapie . . . . . . . . . . . 29
Bildgebung I
V8 Preuhs A, Berger M, Xia Y, Maier A, Hornegger J, Fahrig R:Over-Exposure Correction in CT Using Optimization-BasedMultiple Cylinder Fitting . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 35
-
XVI
V9 Ipek-Ugay S, Tzschätzsch H, Trong MN, Tolxdorff T, Sack I,Braun J: B-Mode-gestützte zeitharmonische Leber-Elastographie zurDiagnose hepatischer Fibrose bei adipösen Patienten . . . . . . . . . . . . . . . 41
V10 Maier A, Kugler P, Lauritsch G, Hornegger J: Discrete Estimationof Data Completeness for 3D Scan Trajectories with Detector Offset 47
V11 Virga S, Dogeanu V, Fallavollita P, Ghotbi R, Navab N, Demirci S:Optimal C-arm Positioning for Aortic Interventions . . . . . . . . . . . . . . . . 53
V12 Lorch B, Berger M, Hornegger J, Maier A: Projection andReconstruction-Based Noise Filtering Methods in Cone Beam CT . . 59
Navigation & Visualisierung
V13 Franz D, Syré L, Paulus D, Bischoff B, Wittenberg T, Hastreiter P:The SIP-NVC-Wizard . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 65
V14 Baer A, Lawonn K, Saalfeld P, Preim B: Statistical Analysis of aQualitative Evaluation on Feature Lines . . . . . . . . . . . . . . . . . . . . . . . . . . . 71
V15 Husch A, Gemmar P, Lohscheller J, Bernard F, Hertel F:Assessment of Electrode Displacement and Deformation withRespect to Pre-Operative Planning in Deep Brain Stimulation . . . . . 77
V16 Saalfeld P, Baer A, Lawonn K, Preim U, Preim B: Das 3D UserInterface zSpace . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 83
V17 Schmitt S, Sobotta C, Choli M, Overhoff HM: Passive 3D NeedleTip Tracking in Freehand MR-Guided Interventions with NeedleModel Visualization . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 89
Registrierung I
V18 Kepp T, Ehrhardt J, Handels H: Evaluation verschiedener Ansätzezur 4D-4D-Registrierung kardiologischer MR-Bilddaten . . . . . . . . . . . . 95
V19 Friedberger A, Museyko O, Engelke K: Binary Image Inpaintingwith Interpolation-Enhanced Diffeomorphic Demons Registration . . 101
V20 Drobny D, Carolus H, Kabus S, Modersitzki J: HandlingNon-Corresponding Regions in Image Registration . . . . . . . . . . . . . . . . . 107
-
XVII
V21 Hering J, Wolf I, Alsady TM, Meinzer H-P, Maier-Hein K: AMemetic Search Scheme for Robust Registration ofDiffusion-Weighted MR Images . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 113
V22 Chen K, Derksen A: A Variational Method for ConstructingUnbiased Atlas with Probabilistic Label Maps . . . . . . . . . . . . . . . . . . . . . 119
Bildgebung (Poster)
P1 Ipek-Ugay S, Drießle T, Ledwig M, Guo J, Tolxdorff T, Sack I,Braun J: MR-Elastographie auf dem Schreibtisch . . . . . . . . . . . . . . . . . . 125
P2 Amrehn M, Maier A, Dennerlein F, Hornegger J: Portability ofTV-Regularized Reconstruction Parameters to Varying Data Sets . . 131
P3 Lu Y, Manhart M, Taubmann O, Zobel T, Yang Q, Choi J-h,Wu M, Doerfler A, Fahrig R, Ren Q, Hornegger J, Maier A:Projection-Based Denoising Method for Photon-CountingEnergy-Resolving Detectors . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 137
Bildrekonstruktion (Poster)
P4 Schober M, Schlömer P, Cremer M, Mohlberg H, Huynh A-M,Schubert N, Kirlangic ME, Amunts K, Axer M: Reference VolumeGeneration for Subsequent 3D Reconstruction of HistologicalSections . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 143
P5 Schubert N, Kirlangic ME, Schober M, Huynh A-M, Amunts K,Zilles K, Axer M: 3D Reconstruction of Histological Rat BrainImages . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 149
P6 Gómez PA, Sperl JI, Sprenger T, Metzler-Baddeley C, Jones DK,Saemann P, Czisch M, Menzel MI, Menze BH: Joint Reconstructionof Multi-Contrast MRI for Multiple Sclerosis Lesion Segmentation . 155
P7 Rempfler M, Schneider M, Ielacqua GD, Xiao X, Stock SR, Klohs J,Székely G, Andres B, Menze BH: Rekonstruktion zerebralerGefässnetzwerke aus in-vivo μMRA mittels physiologischemVorwissen zur lokalen Gefässgeometrie . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 161
P8 Huynh A-M, Kirlangic ME, Schubert N, Schober M, Amunts K,Zilles K, Axer M: Reconstructing a Series of Auto-RadiographicImages in Rat Brains . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 167
-
XVIII
S1 Prinzen M, Trost J, Bergen T, Nowack S, Wittenberg T: 3D ShapeReconstruction of the Esophagus from Gastroscopic Video . . . . . . . . . 173
Navigation & Tracking (Poster)
P9 Scherbinsky M, Lexow GJ, Rau TS, Preim B, Majdani O:Computerunterstützte Planung von Bonebridge Operationen . . . . . . . 179
P10 Alsheakhali M, Yigitsoy M, Eslami A, Navab N: Real Time MedicalInstrument Detection and Tracking in Microsurgery . . . . . . . . . . . . . . . 185
P11 Morariu CA, Terheiden T, Dohle DS, Tsagakis K, Pauli J: EnablingEndovascular Treatment of Type A Dissections . . . . . . . . . . . . . . . . . . . . 191
P12 Sirazitdinova E, Jonas SM, Kochanov D, Lensen J, Houben R,Slijp H, Deserno TM: Outliers in 3D Point Clouds Applied toEfficient Image-Guided Localization . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 197
Registrierung (Poster)
P13 Toso L, Allgeier S, Eberle F, Maier S, Reichert K-M, Köhler B:Iterative Algorithms to Generate Large Scale Mosaic Images . . . . . . . 203
P14 Ehrhardt J, Schmidt-Richberg A, Werner R, Handels H: VariationalRegistration . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 209
P15 Heck C, Benning M, Modersitzki J: Joint Registration andParameter Estimation of T1 Relaxation Times Using Variable FlipAngles . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 215
Physiologische Modellierung (Poster)
P16 Sindel A, Bögel M, Maier A, Fahrig R, Hornegger J, Dörfler A:Respiratory Motion Compensation for C-Arm CT Liver Imaging . . . 221
P17 Antoni S-T, Plagge R, Dürichen R, Schlaefer A: DetectingRespiratory Artifacts from Video Data . . . . . . . . . . . . . . . . . . . . . . . . . . . . 227
-
XIX
Mikroskopie & Optische Verfahren
V23 Stritzel J, Dı́az-Dı́az J, Rahlves M, Majdani O, Ortmaier T,Reithmeier E, Roth B: Korrektur geometrischer Verzeichnungen zurKalibrierung von optischen Kohärenztomographiesystemen . . . . . . . . 233
V24 Liluashvili V, Bergeest J-P, Harder N, Ziesack M, Mutlu A,Bischofs IB, Rohr K: Automatic Single-Cell Segmentation andTracking of Bacterial Cells in Fluorescence Microscopy Images . . . . . 239
V25 Weiss N, Lotz J, Modersitzki J: Multimodal Image Registration inDigital Pathology Using Cell Nuclei Densities . . . . . . . . . . . . . . . . . . . . . . 245
V26 Swarat D, Arens C, Wiemann M, Lipinski H-G: RäumlicheDarstellung und Analyse von Nanopartikelverteilungen in vitalenAlveolarmakrophagen in vitro mit der Dunkelfeldmikroskopie . . . . . . 251
Bildvorverarbeitung & Bildgestützte Dokumentation
V27 Köhler B, Meuschke M, Preim U, Fischbach K, Gutberlet M,Preim B: 2D Plot Visualization of Aortic Vortex Flow in Cardiac 4DPC-MRI Data . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 257
V28 Philipp P, Maleshkova M, Götz M, Weber C, Kämpgen B, Zelzer S,Maier-Hein K, Klauß M, Rettinger A: Automatisierung vonVorverarbeitungsschritten für medizinische Bilddaten mitsemantischen Technologien . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 263
V29 Jose A, Haak D, Jonas SM, Brandenburg V, Deserno TM: TowardsStandardized Wound Imaging . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 269
V30 Haak D, Page CE, Deserno TM: Implementing a Web-BasedArchitecture for DICOM Data Capture in Clinical Trials . . . . . . . . . . 275
Segmentierung II
V31 Egger J, Busse H, Moche M, Brandmaier P, Seider D, Gawlitza M,Strocka S, Garnov N, Fuchs J, Voigt P, Dazinger F, Voglreiter P,Dokter M, Hofmann M, Hann A, Freisleben B, Kahn T,Schmalstieg D: Semi-automatische Segmentierung vonSchädigungszonen in post-interventionellen CT-Daten . . . . . . . . . . . . . 281
-
XX
V32 Nitsch J, Klein J, Miller D, Sure U, Hahn HK: AutomaticSegmentation of the Cerebral Falx and Adjacent Gyri in 2DUltrasound Images . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 287
V33 Rak M, Schnurr A-K, Alpers J, Tönnies K-D: Measurement of theAortic Diameter in Plain Axial Cardiac Cine MRI . . . . . . . . . . . . . . . . . 293
V34 Böer G, Hahmann F, Buhr I, Essig H, Schramm H: Detection ofFacial Landmarks in 3D Face Scans Using the DiscriminativeGeneralized Hough Transform (DGHT) . . . . . . . . . . . . . . . . . . . . . . . . . . . . 299
V35 Morariu CA, Dohle DS, Tsagakis K, Pauli J: Extraction of theAortic Dissection Membrane via Spectral Phase Information . . . . . . . 305
Physiologische Modellierung
V36 Manhart M, Maier A, Hornegger J, Doerfler A: Fast AdaptiveRegularization for Perfusion Parameter Computation . . . . . . . . . . . . . . 311
V37 Wilms M, Fortmeier D, Mastmeyer A, Handels H: ModellbasierteSimulation der Atembewegung für das Virtual-Reality-Training vonPunktionseingriffen . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 317
V38 Bauer S, Keller E, Paulus D: Rückenschmerz durch Übergewicht? . 323
V39 Fischer P, Pohl T, Maier A, Hornegger J: Markov RandomField-Based Layer Separation for Simulated X-Ray Image Sequences 329
V40 Derksen A, Heldmann S, Polzin T, Berkels B: Image Registrationwith Sliding Motion Constraints for 4D CT Motion Correction . . . . . 335
Segmentierung (Poster)
P18 Franz D, Huettmayer H, Stamminger M, Wiesmann V,Wittenberg T: The Cell-Shape-Wizard . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 341
P19 Wang A, Noll M, Wesarg S: Tumorsegmentierung inCD3/CD8-gefärbten Histopathologien . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 347
P20 Jung F, Hilpert J, Wesarg S: Segmentierung von zervikalenLymphknoten in T1-gewichteten MRT-Aufnahmen . . . . . . . . . . . . . . . . 353
-
XXI
P21 Krappe S, Münzenmayer C, Evert A, Koyuncu CF, Cetin E,Haferlach T, Wittenberg T, Held C: Dynamic Programming for theSegmentation of Bone Marrow Cells . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 359
Klassifikation (Poster)
P22 Häfner M, Liedlgruber M, Uhl A: Colonic Polyp Classification inHigh-Definition Video Using Complex Wavelet-Packets . . . . . . . . . . . . 365
P23 Scheel N, Essenwanger A, Münte TF, Heldmann M, Krämer UM,Mamlouk AM: Selection of Seeds for Resting-State fMRI-BasedPrediction of Individual Brain Maturity . . . . . . . . . . . . . . . . . . . . . . . . . . . 371
P24 Morariu CA, Dohle DS, Terheiden T, Tsagakis K, Pauli J: LocalSurface Estimation from Arbitrary 3D Contour Sets for AorticQuantification . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 377
Parallelverarbeitung & Lehre (Poster)
P25 Knopp T: Multithreading-Support für die ProgrammierspracheJulia . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 383
P26 Weber J, Doenitz C, Brawanski A, Palm C: Data-Parallel MRIBrain Segmentation in Clinical Use . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 389
S2 Szalo AE, Zehner A, Palm C: GraphMIC . . . . . . . . . . . . . . . . . . . . . . . . . . 395
Mikroskopie (Poster)
P27 Schetelig D, Wolf IM, Diercks B-P, Fliegert R, Guse AH,Schlaefer A, Werner R: A Modular Framework for Post-Processingand Analysis of Fluorescence Microscopy Image Sequences ofSubcellular Calcium Dynamics . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 401
P28 Schönmeyer R, Brieu N, Schaadt N, Feuerhake F, Schmidt G,Binnig G: Automated Whole Slide Analysis of Differently Stainedand Co-Registered Tissue Sections . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 407
-
XXII
P29 Bier B, Mualla F, Steidl S, Bohr C, Neumann H, Maier A,Hornegger J: Band-Pass Filter Design by Segmentation in FrequencyDomain for Detection of Epithelial Cells in Endomicroscope Images 413
P30 Bug D, Feuerhake F, Merhof D: Foreground Extraction forHistopathological Whole Slide Imaging . . . . . . . . . . . . . . . . . . . . . . . . . . . . 419
Visualisierung (Poster)
P31 Taubmann O, Wetzl J, Lauritsch G, Maier A, Hornegger J: Sharpas a Tack . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 425
P32 Müller A, Knape T, Hufnagl P: GestenbasierteInteraktionsmethoden für die virtuelle Mikroskopie . . . . . . . . . . . . . . . . 431
P33 Koppers S, Schultz T, Merhof D: Spherical Ridgelets forMulti-Diffusion Tensor Refinement . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 437
P34 Meike M, Fortmeier D, Mastmeyer A, Handels H: Real-TimeResampling of Medical Images Based on Deformed TetrahedralStructures for Needle Insertion VR-Simulation . . . . . . . . . . . . . . . . . . . . . 443
S3 Millán-Vaquero RM, Lynch SD, Fleischer B, Rzepecki J, Friese K-I,Hurschler C, Wolter F-E: Enhanced Visualization of the Knee JointFunctional Articulation Based on Helical Axis Method . . . . . . . . . . . . . 449
S4 Prinzen M, Raithel M, Bergen T, Mühldorfer S, Nowack S,Wilhelm D, Wittenberg T: Panorama Mapping of the Esophagusfrom Gastroscopic Video . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 455
Klassifikation & Lernbasierte Verfahren
V41 Gadermayr M, Uhl A, Vécsei A: Dealing with Intra-Class andIntra-Image Variations in Automatic Celiac Disease Diagnosis . . . . . 461
V42 Lüdtke S, Wagner B, Bruder R, Stüber P, Ernst F, Schweikard A,Wissel T: Calibration of Galvanometric Laser Scanners UsingStatistical Learning Methods . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 467
V43 Weber C, Götz M, Binczyck F, Polanska J, Tarnawski R,Bobek-Billewicz B, Meinzer H-P, Stieltjes B, Maier-Hein K:Überwachtes Lernen zur Prädiktion von Tumorwachstum . . . . . . . . . . 473
-
XXIII
V44 Jaremenko C, Maier A, Steidl S, Hornegger J, Oetter N, Knipfer C,Stelzle F, Neumann H: Classification of Confocal LaserEndomicroscopic Images of the Oral Cavity to DistinguishPathological from Healthy Tissue . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 479
V45 Götz M, Weber C, Binczyck F, Polanska J, Tarnawski R,Bobek-Billewicz B, Meinzer H-P, Stieltjes B, Maier-Hein K:Automatische Tumorsegmentierung mit spärlich annotierterLernbasis . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 486
Bildgebung II
V46 Hu S, Riess C, Hornegger J, Fischer P, Bayer F, Weber T,Anton G, Maier A: 3D Tensor Reconstruction in X-Ray Dark-FieldTomography . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 492
V47 Lasser T, Gardiazabal J, Wieczorek M, Matthies P, Vogel J,Frisch B, Navab N: Towards 3D Thyroid Imaging Using RoboticMini Gamma Cameras . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 498
V48 Oppelt MP, Sanders JC, Maier A: Investigation of Single PhotonEmission Computed Tomography Acquired on Helical Trajectories . 504
V49 Möller A, Maaß M, Mertins A: Blind Sparse Motion MRI withLinear Subpixel Interpolation . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 510
V50 Kästner T, Hornegger J, Maier A, Xia Y, Bauer S: TruncationRobust C-Arm CT Reconstruction for Dynamic CollimationAcquisition Schemes . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 516
-
XXIV
Kategorisierung der Beiträge . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 522
Autorenverzeichnis . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 524
Stichwortverzeichnis . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 528
-
Complex Motion Modelling in Cancer Imaging
Julia Schnabel
Department of Engineering Science, University of Oxford
Abstract of Invited Talk
In this talk I will present our recent research efforts and advances in the field ofmotion modelling of complex organs in oncological applications, as part of theCancer Research UK / EPSRC Cancer Imaging Centre at Oxford. I will focuson a number of novel non-linear image registration methodologies developed formotion compensation in single- and multi-modality lung imaging, which is aparticularly challenging application due to the physiological complexity of therespiration process, such as the interaction between rigid structures, interfacingorgans, and large deformations involved. We are currently working on two ma-jor challenges in this field: 1. Correcting for the sliding motion of the lungs,by modelling locally discontinuous deformations, and 2. Formulating computa-tional tractable solutions for image alignment between different types of imagingmodalities.
About the Presenter
Julia Schnabel is Professor in Engineering Science(Medical Imaging) at the Institute of Biomedical En-gineering, University of Oxford, and a Fellow of St.Hilda’s College, Oxford. She received her MSc inComputer Science in 1993 from the Technical Uni-versity of Berlin, and her PhD in Computer Sciencein 1998 from University College London (UCL). Be-fore taking up her academic post at Oxford in 2007,Julia was Research Associate at the Image SciencesInstitute at the University Medical Centre Utrecht,Research Fellow at the Imaging Science Division atKing’s College London and at the Centre for Medi-cal Image Computing at UCL. Julia’s research in herBiomedical Image Analysis (BioMedIA) laboratory focuses on developing in-novative and translational imaging solutions for applications in oncology and
H. Handels et al. (Hrsg.), Bildverarbeitung für die Medizin 2015, Informatik aktuell,DOI 10.1007/978-3-662-46224-9_1, © Springer-Verlag Berlin Heidelberg 2015
-
2 Julia Schnabel
neurosciences. Julia has published over 130 peer-reviewed journal and confer-ence articles, is Associate Editor for IEEE Transactions on Medical Imaging,IEEE Transactions on Biomedical Engineering, and Editorial Board member forMedical Image Analysis.
-
Magnetic Particle Imaging
Chancen und Herausforderungen einer neuen Modalität
Thorsten M. Buzug
Institut für Medizintechnik, Universität zu Lübeck
Kurzfassung
Magnetic Particle Imaging (MPI) ist ein neues Bildgebungsverfahren, mit demsich die lokale Konzentration von magnetischen Nanopartikeln quantitativ so-wohl mit hoher Empfindlichkeit, als auch mit hervorragender räumlicher Auf-lösung in Echtzeit darstellen lässt [1]. Diese Vorteile gegenüber etablierten Ver-fahren, die oft nur einen der Bereiche abdecken können oder nicht quantitativsind, lassen ein hohes klinisches Potenzial in vielen Anwendungen erwarten. DieGrundidee besteht in der Nutzung der nichtlinearen Magnetisierungskurve derPartikel [2]. Das Verfahren nutzt dazu zwei überlagernde Magnetfelder, zumeinen ein statisches Selektionsfeld, zum anderen ein dynamisches Wechselfeld.Werden die Nanopartikel in das Wechselfeld gebracht, erzeugen sie eine nichtli-neare Magnetisierung, die mit einer Empfangsspule gemessen werden kann. Auf-grund der Nichtlinearität enthält das gemessene Signal neben der Grundfrequenzdes Wechselfelds auch Harmonische, also Schwingungen mit einem Vielfachender Grundfrequenz. Nach Separation der Harmonischen von dem eingespeisten
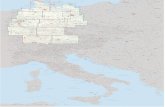
Abb. 1. Simulation (links) und Spulenrealisierung (rechts) eines 2D MPI-Scanners mitdynamisch rotier- und verschiebbarer feldfreier Linie [3].
H. Handels et al. (Hrsg.), Bildverarbeitung für die Medizin 2015, Informatik aktuell,DOI 10.1007/978-3-662-46224-9_2, © Springer-Verlag Berlin Heidelberg 2015
-
4 Thorsten M. Buzug
Grundsignal, kann die Konzentration der Nanopartikel ermittelt werden. Eineörtliche Kodierung wird durch das statische Selektionsfeld erreicht. Als Tracerkommen nanopartikuläre Systeme aus Eisenoxid zum Einsatz. Die Rekonstrukti-on besteht beim Magnetic Particle Imaging in der Lösung des inversen Problems,bei dem zu den gemessenen induzierten Spannungen, die Konzentrationsvertei-lung der Nanopartikel bestimmt werden muss. Die Beziehung zwischen beidenGrößen wird durch eine entsprechende Systemfunktion beschrieben. AktuelleEntwicklungen in der Instrumentierung fokussieren insbesondere auf Spulenop-timierungen [4] (Abb. 1) sowie Konzepte für die Ganzkörpertomographie [5].
1. Buzug TM, Bringout G, Erbe M et al. Magnetic particle imaging: introduc-tion to imaging and hardware realization. Z Med Phys. 2012;22(4):323-34
2. Lüdtke-Buzug K, Haegele J, Biederer S et al. Comparison of commercialiron oxide-based MRI contrast agents with synthesized high-performanceMPI tracers. Biomed Eng. 2013;58(6): 527-33
3. Bente K, Weber M, Gräser M et al. Electronic field free line rotation andrelaxation deconvolution in magnetic particle imaging. IEEE Trans Med Ima-ging. 2014, [Epub ahead of print], DOI: 10.1109/TMI.2014.2364891
4. Wojtczyk H, Bringout G, Tenner W et al. Toward the optimization of D-shaped coils for the use in an open magnetic particle imaging scanner. IEEETrans Magn 2014;50(7):
5. Kaethner C, Ahlborg M, Bringout G et al. Axially elongated field-free pointdata acquisition in magnetic particle imaging. IEEE Trans Med Imaging.2015;34(2):1-7
Über den Vortragenden
Prof. Buzug promovierte 1993 im Fach AngewandtePhysik an der Universität zu Kiel. Nach einer postdok-toralen Position an der Forschungsanstalt der Bun-deswehr für Wasserschall- und Geophysik (FWG) inKiel, wo er im Bereich der Unterwasserbildgebung anSONAR-Systemen arbeitete, wechselte er 1994 zu denPhilips Forschungslaboratorien Hamburg. Als Leiterdes Forschungsclusters Bildverarbeitung war Prof. Bu-zug dort für Projekte der medizinischen Bildverarbei-tung verantwortlich. Prof. Buzug wurde 1998 auf eineC3-Professur für Physik und Medizintechnik an denRheinAhrCampus Remagen berufen. 2006 wurde erin seine derzeitige Position als Direktor des Instituts für Medizintechnik an derUniversität zu Lübeck berufen. Prof. Buzug ist unter anderem Vizepräsidentder Universität zu Lübeck, Sprecher des Kompetenzzentrums für Medizintech-nik TANDEM und Vorstandsmitglied des Life-Science Nord e.V. (LSN).
-
Data-Driven Spine Detection for Multi-SequenceMRI
Daniel Kottke1, Gino Gulamhussene1, Klaus Tönnies2
1Otto-von-Guericke-University Magdeburg (equal contributors)2Computer Vision Group, Otto-von-Guericke-University Magdeburg
Abstract. Epidemiology studies on vertebra’s shape and appearancerequire big databases of medical images and image processing methods,that are robust against deformation and noise. This work presents asolution of the first step: the vertebrae detection. We propose a methodthat automatically detects the central spinal curve with 3D data-drivenmethods in multi-sequence magnetic resonance images (MRI). Addition-ally, we use simple edge operations for vertebra border detection thatcan be used for a statistical evaluation with help of some fast user inter-action. Our automatic vertebrae detection algorithm fits a polynomialcurve through the spinal canal, that afterwards is shifted towards thevertebra centers. An edge operator gives a first approximation of thevertebra borders, that can be evaluated and corrected by some user in-teraction within 12 seconds. We show, that our algorithm automaticallydetects more than 90% of all spines correctly, and present a preliminaryanalysis of vertebrae sizes.
1 Introduction
Huge field studies using medical images are a trade-off between the numberof participants and the cost of each. Normally, computer science studies onmagnetic resonance imaging (MRI) consist of a small number of subjects, andhigh quality images. The “Study of Health in Pomerania” (SHIP) [1] producedapproximately 3300 datasets of human spine images. In contrast to many otherstudies, the number of available data is huge and enables statistical analysis ofhuman anatomy in the normal population. However, the image sampling ratewas reduced which complicates the image processing task.
Studying the anatomy of spine and vertebrae and thus generating anatomicalnorms for the normal population is one of the goals of SHIP. This work proposesa spine detection algorithm and gives an outlook on vertebra identification. Themerit of our method lies in its data-driven nature, which ensures robustness to-wards vertebrae deformation and prevents model induced biases. The automaticprocess is divided into three phases: spinal canal extraction, spine curve calcu-lation and vertebra border detection. Furthermore, we provide a graphical userinterface, that allows validation and correction with fast user interaction.
Source code is available at: https://bitbucket.org/dkottke/parsos
H. Handels et al. (Hrsg.), Bildverarbeitung für die Medizin 2015, Informatik aktuell,DOI 10.1007/978-3-662-46224-9_3, © Springer-Verlag Berlin Heidelberg 2015
-
6 Kottke, Gulamhussene & Tönnies
The most challenging problem on medical images is the image quality. Hence,research on spinal detection and segmentation relies on top-down (model-based)methods. To cope with vertebrae deformation, one can use model descriptions(e.g. vertebra size ratios) or capture representative variations in a complex model.We observed that researchers preferred using more complex models in recentyears. Specialized models only detect parts of the spine [2, 3, 4, 5, 6] whereasothers use the full spine [7, 8]. 3D methods (e.g. [2, 4, 5]) allow more complexmodels.
One recent paper on segmentation of vertebrae in CT images was proposedby Kadoury et al. [3]. They segmented 12 thoracic and 5 lumbar vertebralbodies (VB) with help of a previously learned articulated deformable model andhigher order markov random fields. MRI based algorithms rely on model-basedsegmentation and detection i.e. statistical shape models [4, 8], or graphicalmodels [5]. Other methods build complex learning algorithms, e.g., Huang etal. [7] used an AdaBoost algorithm on wavelet transformed vertebrae, followedby a normalized cut segmentation. Some algorithms require initial user input,as they need key markers for an approximate detection [3, 5]. Others replacedthis step by locating a central line through the spine or the spinal canal [4,9, 7]. Neubert et al. [4] and Stern et al. [9] use a method based on the workby Vrtovec [10]. Here, a curve through the vertebra centers defines a localcoordinate system of the spine, that facilitate to transform the whole spine intoa straight one with more simple properties. Pohle-Frölich et al. [6] uses symmetrytransformation to detect the spine position.
The next section provides a detailed description of our method in Sec. 2. InSec. 3, we discuss our results and finally conclude our work in Sec. 4.
2 Materials and methods
Our data comprises 3D registered T1 and T2 weighted MR images of 3300 sub-jects that took part in the “Study of Health in Pomerania” (SHIP) [1]. Theimages cover the whole spine in 15 sagittal slices (1.11 mm x 1.11 mm x 4.4 mm)and contain a marker line in the center that locates the slice where the upperspine image was connected to the corresponding lower part. This is recognizedand removed because we process the complete spine.
Our method does not use a spine model and consists of five phases (Fig. 2).First, we generate a mask for the spinal canal region that is used as a globalregion of interest (ROI) for all subjects. The main part contains three automaticphases: the extraction of the spinal canal using the pre-calculated ROI, thecalculation of the polynomial through the vertebra centers (spine curve), andthe detection of the vertebra borders. The fifth phase is interactive and ensurescorrect results with help of a graphical user interface.
2.1 Mask generation
To create a global mask, we manually segmented the cerebrospinal fluid from 10randomly selected subjects. Due to varying body positions and shapes among
-
Data-Driven MRI Spine Detection 7
the subjects, we did not use the absolute coordinates of the segmentation. Eachtransverse image plane was shifted such that the back was moved to the imageborder. We used the logical OR operator to combine the different transformedmasks, that finally are dilated separately for every spine part. The dilationdegree depends on the variance between the different segmentations. Thus, wehave a mask that defines the region of interest relative to the subject’s backposition.
2.2 Spinal canal extraction
To find the spinal canal, we used the mask from the previous step. It is placedrelatively to the subject’s back, which is localized by an intensity comparison,as the rightmost visible structure in our MR images (Fig. 2 (a)). This regionof interest (ROI) ensures that structures i.e. fluids, that are far from the spine,will not be considered. To create an estimate of the bias field, we used a max-imum filter with infinite kernel depth and width and a height of 16mm. Eachsequence (T1 and T2-weighted MRI) is multiplied with the inverse of the biasfield estimate to remove this artifact. Since cerebrospinal fluid appears dark inT1 and bright in T2 weighted MR images, subtracting the T1 weighted fromthe T2 weighted image, generates an image where only fluids are bright. Tofind tubelike structures these images are convolved with kernels filled with onesof 20x2x2mm size and ±15◦ and ±30◦ orientation. To detect outliers, we useinformation of volume and variance of each connected segment in the binaryimage after thresholding. Fitting a polynomial curve on the means of the axialslice, we produce a non-linear transformation such as in Vrtovec [10] (Fig. 2 (b)).This simplifies the future operations: the detection of the central spinal curveand vertebrae borders.
2.3 Spine curve calculation
The polynomial curve from the previous step can be used to create a curvedplanar reconstruction (CPR) [10]. This provides a straight spinal canal and aapproximate straight spine (Fig. 2 (c)), which simplifies subsequent steps.
Fig. 1. Graphical summary of the complete algorithm.
-
8 Kottke, Gulamhussene & Tönnies
Since vertebral bodies, intervertebral discs and other neighboring structurescannot be distinguished only by intensity, the most valuable information are theedges between vertebral bodies and intervertebral discs. Near the spine centers([−8mm, 8mm]) these edges do not vary in the sagittal plane because of theCPR. Summing the derivatives of the image slices sagittally produces a noisereduced edge image (Fig. 2 (c) left). Using a percentile filter with a kernel thatoverlaps at least one vertebra edge and smoothing the result with a Gaussianfilter, we get an image with the highest values along the centerline of the spine(Fig. 2 (c) center and right). Tracking the local maximums produces the shiftedpolynomial.
2.4 Vertebrae border detection
To detect the borders between vertebral bodies and intervertebral discs, wesearch for significant edges. In order to minimize the noise we add the firstderivative of both T1 and T2 weighted MR images pixel-wise. This is equivalentto the mean derivative of both images. Then, we distinguish between strongrising (start of a vertebral body (VB)) and falling edges (end of VB) with a zerocrossing at the second derivative of the mean image. Hence, we can generate afirst estimation of vertebral body borders by assigning starting and ending edgesaccording to their order. In an iterative process we eliminate inconsistencies byassuming that the vertebral body length and the intervertebral disc thicknesschange linearly with its position on the spine. We estimate this model iterativelyby linear regression and distinguish outlying data points. When segments are ab-normally large, additional edges are searched, and edges are deleted if segmentsare abnormally small. The process ends when no more outliers are detected.
2.5 Manual post processing
In order to assess the results, we implemented a graphical user interface, provid-ing a 2D sagittal view of the spine with its polynomial curve and the detectedvertebrae with their borders. Furthermore, this tool allows the user to removewrongly detected vertebrae and add missing ones, while being able to zoom andslide through the sagittal slices.
Fig. 2. With help of the mask a regionof interest (ROI) is defined for the rawT1 and T2 weighted images (a) in whicha polynomial is fitted to the spinal canal(b). This is used for a curved planar re-construction (CPR) which is convolvedwith a percentile filter and a Gaussian fil-ter afterwards to find the vertebrae cen-terline (c).