Biotechnologie - bilder.buecher.de · mid pSC 101 (Abb. 3.29), das in hoher Zahl in der Zelle...
Transcript of Biotechnologie - bilder.buecher.de · mid pSC 101 (Abb. 3.29), das in hoher Zahl in der Zelle...
� 3.11 Molekulare Scheren und
Kleber: Restriktionsendonucleasen
und DNA-Ligasen
In den 60er-Jahren hatten Werner Arber (geb.1929) (Abb. 3.28) in Genf und der AmerikanerHamilton D. Smith (geb. 1931, beide bekamen1978 den Nobelpreis mit Daniel Nathans, 1928 -1999) einen Schutzmechanismus der Bakteriengegen die tödliche Bedrohung durch Bakterienvi-ren (Bakteriophagen) entdeckt. Bakteriophageninjizieren ihre DNA in die Bakterienzellen. Die Bak-terien zerschneiden die fremde Virus-DNA mitEnzymen, so genannten Restriktionsendonu-cleasen (kurz: Restrictasen), und machen siedamit unschädlich. Die bakterieneigene DNA wardagegen durch zusätzliche Methyl-(CH3- )Gruppengeschützt, welche die Restrictasen blockieren.
1970 fand man heraus, dass Restriktionsendonu-cleasen die DNA nicht beliebig, sondern nur anganz bestimmten Basenpaaren exakt zerschneiden.Herbert W. Boyer (geb. 1936, Abb. 3.24) unter-suchte an der Universität von Kalifornien in SanFrancisco die Restriktionsendonuclease EcoRI(benannt nach dem Escherichia coli -Stamm RY13). Sie zerschneidet DNA nur dort, wo die Basen-kombination „GAATTC“ auftritt und zwar zwi-
schen den Basen G und A. An dem gegenüberlie-genden komplementären „Schwester“-DNA-Strang mit der Basenfolge „CTTAAG“ spaltet Eco-RI ebenfalls zwischen den Basen A und G:
3’-XXXXXXXXG/AATTCXXXXXXXX-5’
5’-XXXXXXXXCTTAA/GXXXXXXXX-3’
So entsteht kein „glatter Schnitt“, sondern es bildensich zwei Bruchstücke mit überstehenden Enden:
3’-XXXXXXXXG AATTCXXXXXXXX-5’
5’-XXXXXXXXCTTAA GXXXXXXXX-3’
Doch die zerschnittene DNA zerfällt bei niedrigerTemperatur nicht in zwei Teile, ihre überstehendenEnden kleben lose aneinander. Janet Mertz aus PaulBergs (Abb. 3.47) Labor an der Stanford Universitätfand heraus, dass sich die A- und T- sowie C- und G-Basen, diese „sticky ends“ (klebrige Enden),elektrostatisch anziehen. Man kann sie sogar wie-der durch ein Enzym, die ATP verbrauchendeDNA-Ligase, zusammenfügen.
Voneinander unabhängig suchende Forscher hat-ten „Scheren“ und „Kleber“ für die DNA gefunden,nun mussten die Ergebnisse ihrer Anstrengungenvereint werden.
Heute sind über 1200 Restriktionsendonucleasenbekannt, die drei verschiedene Klassen bilden. Nureinige von ihnen sind allerdings interessant für dieGentechnik. Es gibt übrigens auch Restriktionsen-donucleasen mit glattem Schnitt und glatten Enden(blunt ends), z.B. Pvu II aus Proteus vulgaris undAlu I aus Arthrobacter luteus.
Verschiedene Enzym-Scheren erzeugen gleicheEnden: Die Restriktionsendonucleasen Bam HI(Erkennungssequenz GGATCC) aus Bacillus amy-loliquefaciens und Bgl II (ErkennungssequenzAGATCT) aus Bacillus globigii lassen beide klebri-ge Enden mit der gleichen Sequenz GATC entste-hen. Die Genfragmente, die von einem dieser Enzy-me produziert wurden, lassen sich also mit denendes anderen verbinden.
� 3.12 Die ersten Gentechnik-
experimente: Quakende Bakterien?
Anfang 1973, knapp ein Jahr, nachdem die Werk-zeuge verfügbar waren, führten Stanley N. Cohenund seine Mitarbeiterin Annie C. Y. Chang von derUniversität Stanford mit ihren Kollegen der benach-barten Universität von San Francisco, Herbert W.Boyer (Abb. 3.24) und Robert H. Helling, das ersteExperiment der neuen Gentechnologie aus. DieForscher wählten das nach den Initialen von Stan-ley Cohen benannte kleine nichttransferable Plas-
Biotechnologie
62
Abb. 3.28 Werner Arber(geb. 1929)
Abb. 3.29 Das PlasmidpSC101, von Stanley Cohenauch plasmid necklace (Plas-mid-Halskette) genannt
Abb. 3.27 Das erste Gen-technikexperiment vonCohen und Boyer
✂✂
Gen fürTetracyclinresistenz
pSC102
Spalten mitEcoRI
pSC101
Gen fürKanamycinresistenz
Hybridplasmid
C
TT A A
G
G
AA T T
C
Tc Spalten mitEcoRI
Verknüpfungmit Ligasen
Tc
Ka
✂
✂
GA
AT
TC
CT
TA
A
G
✂
✂
✂
Ka
Ka
Verknüpfungmit Ligasen
Tc
K
CT
T
AA
G
G AA
TT
C
mid pSC 101 (Abb. 3.29), das in hoher Zahl in derZelle vorliegt. Es trägt ein Gen, das tc-Gen, das E.coli resistent gegen das Antibiotikum Tetracyclinmacht. Das Plasmid pSC 101 wurde ausgewählt,weil es nur eine einzige Basenanordnung enthält, dievon der Restriktionsendonuclease EcoRI zwischen Gund A in der Sequenz -GAATTC- gespalten wird.Wenn es mehrere Schnittstellen gegeben hätte, wür-de das ringförmige Plasmid nicht einfach aufge-schnitten, sondern in viele Stücke geteilt werden.
Die Fähigkeit zur Antibiotikaresistenz durfte durchdas Aufschneiden keinesfalls verloren gehen. Manwollte ja später herausfinden, in welche Bakterien-zellen das fremde Gen erfolgreich eingeschleustworden war. EcoRI stammt – wie der Name sagt –aus Escherichia coli. Durch EcoRI wird die ringför-mige Plasmid-DNA in fadenförmige lineare DNAmit klebrigen Enden verwandelt. Cohen und Boyerzerschnitten mit EcoRI auch ein anderes Plasmidaus E. coli (pSC 102), das ein Gen für die Resistenzgegen das Antibiotikum Kanamycin (Ka-Gen)enthält (Abb. 3.27). Auch dieses Plasmid hatte nureine Schnittstelle und das Ka-Gen wurde von Eco-RI nicht zerschnitten.
Die zwei zerschnittenen Plasmide besaßen die glei-chen klebrigen Enden, da sie an der gleichen Stelle-G/AATTC- zerschnitten worden waren. Durch dieelektrostatischen Anziehungskräfte zwischen denBruchstellen lagerten sich die zwei unterschiedli-chen DNA-Bruchstücke lose zusammen. Die Wis-senschaftler fügten nun den Kleber, DNA-Ligase(Abb. 3.33), dem Gemisch hinzu und verbandendamit die zwei Klebestellen. Es entstanden neuegrößere rekombinante Plasmide.
Im letzten Schritt, der Transformation, wurdediese rekombinante DNA in Bakterien überführt.Dazu wurde der Lösung mit den zu manipulieren-den E. coli -Bakterien Calciumchlorid (CaCl2) zuge-setzt. Dieses Salz macht die Zellwände für DNAdurchlässig (Abb. 3.30). Mit diesem künstlichenVorgang, der in der Natur so nicht vorkommt,konnten die neuen Plasmide in die Bakterien einge-schleust werden (siehe auch Abb. 3.23). Am Schlussfolgte der entscheidende Test: Die Bakterienlösungwurde auf Nährplatten ausgestrichen, die sowohlTetracyclin als auch Kanamycin enthielten.
Die meisten Bakterien gingen – wie erwartet – ein.Nur wenige Bakterien überlebten. Sie mussten daskünstlich geschaffene Plasmid mit der Doppelre-sistenz besitzen: Hier wurden Kanamycin undTetracyclin desaktivierende Enzyme von den Bak-terien gebildet. Die überlebenden Bakterien ver-mehrten sich und wuchsen zu Zellkolonien heran;
etwa 100 Millionen identisch gebauter Nachkom-men, die alle die neue rekombinante DNA trugen.Ein Klon war entstanden, eine Gruppe genetischidentischer Lebewesen.
Nach diesem Erfolg wagten sich Cohen und seineMitarbeiter an das nächste Experiment. Sie rekom-binierten DNA-Segmente von Plasmiden aus ver-schiedenen Bakterien, aus E. coli das Plasmid pSC101 und ein Plasmid aus dem penicillinresistentenStaphylococcus aureus. Da das Plasmid von St.aureus vier Schnittstellen für EcoRI enthielt, wurde
Die Wunder der Gentechnik
63
Spaltung mitRestriktions-
endonucleasen
DNA mit gesuchtem Gen
DNA-Fragment mitgesuchtem Gen
andere DNA-FragmenteSpaltung von Plasmidenmit gleichen Restriktions-
endonucleasen
Mischen und Verknüpfen mit Ligasen
Einschleusen in Bakterien
Kultivierung, Koloniebildung
Waschen
Übertragung auf Filterpapier,Aufschluss der Bakterien
radioaktiv markierte mRNAdes gesuchten Gens
Schwärzung einer Fotoschichtzeigt radioaktiv markierte
mRNA vom gesuchtem Gen
Hybridisierung
Identifizierungdes gesuchten Klons
CaCl2
Abb. 3.30 Prinzip der Klonie-rung eines Gens aus Säuger-zellen
Was kann man aus Genen
herauslesen?
»Gene sind nicht mit Blau-pausen für Ingenieure zu ver-gleichen, eher mit Rezeptenin einem Kochbuch. Siesagen uns, was für Zutatenreinkommen, in welchenMengen und welcher Reihen-folge – doch sie liefern unskeinen vollständigen undgenauen Plan dessen, wasdabei herauskommt.«
Ian Stewart, Life’s OtherSecret, New York 1998
Box 3.2 Nützliche Vektoren,
Transportmittel für Gene
Das Plasmid pBR322
Das bei Gentechnikern sehr beliebte PlasmidpBR322 aus E. coli wurde Ende der 70er-Jahre entwickelt. p bedeutet Plasmid, BR be-zeichnet die beteiligten Wissenschaftler Boli-var und Rodriguez. Die Zahl 322 unterschei-det das Plasmid von anderen Plamiden ausdem gleichen Labor wie pBR325, pBR327usw.
pBR322 enthält Gene für die Antibiotikare-sistenz-Enzyme gegen Tetracyclin und Ampi-cillin. Verschiedene Restriktionsendonuclea-sen können das Plasmid an spezifischenSchnittstellen aufschneiden. Anschließendlassen sich dort ebenso geschnittene DNA-Fragmente einfügen. Wenn Eco RI benutztwird, wird keines der Antibiotikaresistenzge-ne zerschnitten, wohl aber, wenn z. B. mitBam HI geschnitten wird. Dann ist dasTetracyclinresistenzgen zerschnitten, und eswird ein Fremdgen mitten in das tc-Gen ein-gebaut (Insertion).
Die Zellen sind dann resistent gegen Ampi-cillin, aber nicht gegen Tetracyclin und kön-nen so leicht selektiert werden.
Zellen, die den Vektor nicht aufgenommenhaben, sind gegen beide Antibiotika emp-findlich. Zellen, die pBR322 ohne Insertionenthalten, sind gegen beide Antibiotika resis-tent.
Der λ-Phage
Der λ-Phage (Phage Lambda) liebt Abwechs-lung: Er kann seinen Wirt zerstören oder
vorübergehend ein Teil von ihm werden. Imersten Fall werden bei der Lyse virale Protei-ne und DNA schnell erzeugt und zu Virus-partikeln verpackt, was zur Zerstörung derWirtszelle führt (lytischer Zyklus). Imzweiten Fall, dem lysogenen Zyklus, fügtsich die Virus-DNA in das Genom der Wirts-zelle ein. Sie wird mit deren DNA eventuellüber Generationen hinweg repliziert, ohnedem Wirt zu schaden.
Durch bestimmte Umweltänderungen kanndie ruhende Virus-DNA plötzlich aktiviertwerden. Sie wird aus dem Genom herausge-schnitten und initiiert den lytischen Zyklus.Der Phage λ hat 48 kb DNA, und große Tei-le sind für eine erfolgreiche Infektion nichtnotwendig. Gene für den lysogenen Zykluskönnen also durch fremde DNA ersetzt wer-den, ideal für einen Vektor.
Für die Klonierung von DNA wurden gezieltPhagen-Mutanten entwickelt. Deren DNAkann nur an zwei Stellen (anstelle von fünf)durch EcoRI geschnitten werden. Dabei ent-stehen drei Schnittprodukte, von denen dasmittlere entfernt wird.
An seine Stelle wird ein passendes langesDNA-Stück (etwa zehn kb) mit Ligasen ein-gefügt. Der Phage ist dadurch noch immerinfektiös, kann aber nur den lytischen undnicht den lysogenen Zyklus durchlaufen(also nicht „schlafend abwarten“). Das sindalles sehr wünschenswerte Eigenschaften füreinen Klonierungsvektor. Der Vorteil vomPhagen λ ist, dass die manipulierten Virenleichter als Plasmide in Bakterienzellen ein-dringen und größere DNA-Abschnitte ein-führen können.
Man hat auch spezielle Cosmide konstru-iert. Sie sind Hybride aus λ-Phagen und Plas-miden. Der Name Cosmid leitet sich vonDNA-Sequenzen ab, die als cos bezeichnetwerden und von dem Bakteriophagen λstammen.
Diese Abschnitte machen es möglich, dass inCosmide größere Gene (bis zu 45 kb) einge-baut werden können. Solche Cosmide wer-den in Phagen verpackt, mit deren Hilfe sichletztlich die fremden Gene in Bakterien ein-schleusen lassen.
Enthalten die Cosmide das Gen für dieAmpicillinresistenz, werden die Bakterien indie Lage versetzt, in einer Kulturlösung trotzZugabe des Antibiotikums Ampicillin zuüberleben und sich und die Fremd-DNA zuvermehren.
Der M13-Phage
M13 ist ein filamentöser Phage. Er sieht völ-lig anders aus als der Phage λ. Es handeltsich um ein fadenförmiges Virus (900 nmlang, mit nur neun nm Durchmesser) miteiner ringförmigen einzelsträngigen DNA(auch +-Strang genannt) und einer Protein-hülle aus 2 710 identischen Proteinunterein-heiten (siehe Kap. 5).
Das Virus dringt in E. coli interessanterwei-se über deren „Sexualorgan“ ein, den Sexpi-lus, der den Austausch von DNA zwischenBakterien ermöglicht. M13 ist so attraktiv,weil man mit ihm die klonierte DNA in ein-zelsträngiger Form gewinnen kann. Einzel-stränge von klonierten Genen braucht manbesonders für die DNA-Sequenzierung unddie in vitro-Mutagenese (Kap. 10).
M13 ist der Akteur bei der Phagen-Dis-play-Technik (Kapitel 5). Die M13-DNAwird nicht (wie beim Phagen λ) in das Bak-teriengenom integriert. Es kommt auchnicht zur Lyse der Zellen. Das Bakteriumwächst und teilt sich, allerdings langsamerals nichtinfizierte Zellen. Die Tochterzellensetzen weiterhin M13 Phagen frei.
Pro Generation entstehen etwa tausendneue M13-Phagen. Da M13 den Wirt nichttötet, kann man ihn in großen Mengenzüchten und leicht ernten.
Biotechnologie
64
Pst I
Pvu I
Sal I
BamH I
EcoR I Hind III
amp tc
ori
4363 bp
Scal I
Die genetische und physikalische Karte vonpBR322 zeigt die Positionen der zwei Resistenz-gene für Ampicillin (amp) und Tetracyclin (tc),den Startpunkt der Replikation (ori) undSchnittstellen wichtiger Restriktionsendo-nucleasen.
Der λ-Phage
Der Phage M13 ist ein filamentöses Virus, das heißt langgestreckt
es folglich in vier Teile zerstückelt. Nur ein DNA-Stückchen enthielt dabei das Resistenzgen gegenPenicillin. Die verschiedenen Kombinationsmög-lichkeiten waren somit größer als im ersten Experi-ment. Die Stunde der Wahrheit kam dann mit derAusplattierung der manipulierten Bakterien auftetracyclin- und gleichzeitig auch penicillinhaltigenNährboden.
Ein Jubelschrei aus Cohens Labor: Auch ein solchesrekombinantes Plasmid funktionierte in den E. coli -Zellen. Damit wurde erstmals Erbmaterial unter-schiedlicher Arten verpflanzt. Eine „Überwindungder Schranken, die normalerweise biologischeArten voneinander trennen“, wie Cohen etwas vor-eilig verkündet hatte, war allerdings nicht erreichtworden, weil – wie wir heute wissen – auch unter-schiedliche Mikroorganismen und Viren Erbmateri-al „natürlich“ austauschen können. Alle Virusin-fektionen sind also Gentransfer!
Ermutigt durch diese Erfolge, sollten nun höhereBarrieren überwunden werden: die Artschrankezwischen Bakterien und Fröschen, genauer denAfrikanischen Krallenfröschen (Xenopus lae-vis) (Abb. 3.32).
Genomische DNA wurde aus Krallenfroschzellenisoliert und mit Restriktionsenzymen des TypsEcoRI zerschnitten. Gleichzeitig wurde das Bakte-rienplasmid pSC 101 ebenfalls mit EcoRI aufge-schnitten (Abb. 3.31).
Zusammengelagerte Frosch-DNA und Bakterien-DNA wurden mit Ligasen verklebt, in E. coli -Zelleneingeschleust und vermehrt. Die Zellen, die das
neue rekombinante Frosch-Bakterien-Plasmid ent-hielten, wurden aufgrund der Tetracyclinresistenzund – da die Frosch-DNA keine Resistenzgenegegen Antibiotika enthält – durch chemische Ana-lyse der Nucleinsäuren identifiziert.
Am 27. Juli 1973 stand fest: Frosch-DNA wird vonBakterien „akzeptiert“!
Das neue Plasmid vermehrte sich 1000-fachen beiden 1000 Zellteilungen mit. Von ihm wurden somit identische Kopien hergestellt: Das rekombinanteFrosch-Bakterien-Plasmid wurde kloniert.
Eine neue Art war das noch nicht, denn nur weni-ger als ein tausendstel der Bakterien-DNA stammtvom Frosch. Zwar quakten die manipulierten Bak-terien nicht nach Froschart, wie die Forscherscherzten, aber etwas viel Wichtigeres war von denersten Gentechnikern geleistet worden: Sie hatteneine universelle Gentechnik-Methode entwickelt,mit der erstmals das bis dahin völlig unzugänglicheErbmaterial höherer Lebewesen in großer Mengehergestellt und untersucht werden konnte: dasKlonieren von DNA war nun technisch möglichgeworden (Abb. 3.30).
� 3.13 Wie Gene gewonnen werden
Leider war das Experiment mit der „zerhackten“Frosch-DNA nicht so einfach auf die Produktionvon Proteinen höherer Lebewesen zu übertragen.Der Grund dafür waren die in die DNA-Kette ein-gefügten Introns mit ihrer nicht Protein codieren-den „Nonsense“-Information. Es hätte nicht vielSinn, die DNA höherer eukaryotischer Lebewesendurch Restriktionsendonucleasen zu spalten und in
Die Wunder der Gentechnik
65
Abb. 3.32 Afrikanischer Krallenfrosch (Xenopus lae-
vis) – ein neues Labortier wiezuvor Maus und Ratte, an-spruchslos und langlebig.Sein wissenschaftlicherName („seltsamer Fuß“)kommt von den schwarzenKrallen. In Kalifornien wur-den diese fresslustigen, überzehn Zentimeter großen ein-gebürgerten Amphibien zurLandplage.
Plasmid
Ligase
Spaltenmit EcoRI
Kultur von E. coli mitTetracyclinresistenz (tc)
„klebrige Enden“
LigasLigaseLigas
CaCl2
Einschleusen in E. coliSelektion dertetracyclinresistentenKlone
Hybridplasmid
angereicherte DNAKrallenfrosch
„klebrige Enden“
Gen fürTetracyclinresistenz
tc
tc
tc
Gen fürTetracyclinresistenz
Ligase
Spaltenmit EcoRI
Abb. 3.33 DNA Ligase, der„Kleber“ für geschnitteneDNA, ist ein ATP-abhängigesEnzym (siehe Kap. 2). Ligaseist ein für die Gentechnikunverzichtbares Werkzeug.Der Cofaktor ATP (rot) undein Lysin-Rest (magenta)sind essenziell.
Abb. 3.31 Einbau und Klonierung von Krallen-frosch-DNA in Bakterien
Plasmid-DNA einzubauen. Zwar könnten Tausen-de von Fremd-DNA-Stücken kloniert werden, aberein funktionierendes Fremdprotein würde kaumproduziert. Im besten Fall wäre das Endprodukt einProtein, das zwar alle seine Aminosäuren aus denExons enthält, dazwischen aber völlig irrelevante„Extra-Aminosäuren“ aus den Introns.
Der Ausweg für die Gentechniker lag darin, nichtdie intronbelastete DNA höherer Lebewesen zuverwenden, sondern die reife (mature) mRNA zugewinnen, auf der die verschlüsselten Baupläne derProteine von Introns befreit sind. Nun lässt sichaber die einzelsträngige mRNA nicht mit der dop-pelsträngigen Plasmid-DNA zusammenbauen. Glück-licherweise fand man (wie bereits in Abschnitt 3.3erwähnt) ein Enzym in Retroviren, die reverseTranskriptase (Revertase). Sie kann die einzel-
strängige RNA in doppelsträngige DNA zurück-schreiben (Abb. 3.35). Die Erbsubstanz der Retro-viren besteht nämlich nicht aus DNA, sondern auseinem einzelsträngigen RNA-Molekül. Befallen die-se Viren DNA enthaltende Wirtszellen (siehe Kapi-tel 5), übersetzen sie mit der mitgebrachten Rever-tase ihre einzelsträngige Viren-RNA in doppelsträn-gige DNA und integrieren diese dann in die Wirts-DNA (Abb. 3.23).
Die Gentechniker nutzen nun reverse Transkripta-se, um an dem mRNA-Einzelstrang einen DNA-Strang zu synthetisieren. Diese DNA, die durchKopieren einer RNA entstanden ist, wird als copy -DNA (cDNA) bezeichnet.
Ist die Abfolge der Aminosäuren (Sequenz) einesProteins bekannt und lässt sich die entsprechendemRNA dennoch nicht aus Zellen isolieren, kann daszugehörige Gen aber auch auf chemischem Wegsynthetisiert werden. So lässt sich eine DNA her-stellen, die eigentlich in der Natur nicht vorkommt.Heute gibt es schon DNA-Syntheseautomaten(Box 3.5). Bei ihnen wird ein Start-Nucleotid an fes-te Trägermaterialien (wie Silicagel oder Glasperlen)gebunden und nachfolgend hängt man Nucleotidein der gewünschten Reihenfolge an. 1988 konnteman so täglich 30 Basen lange DNA-Fragmente syn-thetisieren: Dazu war noch 1979 ein halbes JahrTeamarbeit nötig. Heute kann man in wenigen Stun-den komplette „Gene nach Maß“ synthetisieren.
� 3.14 Humaninsulin aus Bakterien?
Im Juli 1980 erhielten 17 Freiwillige Insulininjek-tionen im Londoner Guy’s Hospital. Sie machtenSchlagzeilen in den Zeitungen. Was war daran sosensationell? Jeden Tag wurden Millionen Diabeti-ker in aller Welt mit dem Hormon Insulin behandelt(siehe Box 3.3). Das Insulin wurde aus der Bauch-speicheldrüse von Rindern und Schweinen gewon-nen. Das substituierte Insulin soll den Blutzucker-spiegel der Diabetiker regulieren und die schwer-wiegenden Folgen des Diabetes bekämpfen – eineKrankheit, die in den Industrieländern auf Platzdrei der Todesursachen steht (siehe Box 3.4).
Der Typ-I-Diabetes wird durch Autoimmunzerstö-rung der Insulin bildenden Zellen des Pankreas ver-ursacht und setzt vor dem 20. Lebensjahr ein. DerTyp-II-Diabetes tritt dagegen ab mittlerem Lebens-alter auf (90 % der Diabetiker), vor allem bei Über-gewichtigen (Kap. 10). Die oben erwähnten 17Freiwilligen waren die ersten Menschen in derMedizingeschichte, die mit einem Säugetierhor-mon behandelt wurden, das nicht aus Säugetieror-
Biotechnologie
66
Abb. 3.34 Synthese von Ratten-Proinsulin durch Bakterien
Schneiden mitRestrictase
Einbau desProinsulin-
Gens
Einschleusen in Bakterien,Genexpression
Gen fürTetracyclin-resistenz
Penicillin-resistenz-Gen
Mischprotein
Teil des Penicillinasemoleküls
Proinsulin
aktives Ratteninsulin
Trypsin-behandlung
Teil desPenicillin-resistenz-Gens
Tc
Abb. 3.35 Unten: ReverseTranskriptase. Ihre klauenartige Struktur.Darunten: Zwei Aktivitätensind in einem Molekül derreversen Transkriptase ver-eint: Mit der Polymerase ent-steht ein RNA-DNA-Hybrid-molekül, mit der Nuclease-Aktivität wird die am Endeüberflüssige RNA abgebaut.Der DNA-Einzelstrang wirddann zum Doppelstrangergänzt.
Polymerase
Nuclease
Die Wunder der Gentechnik
67
Abb. 3.36 Insulinsynthese in den Inselzellen des Pankreas:
Gezeigt ist ein typischesInsulingen einer Säugetier-zelle mit Introns, codieren-den Sequenzen (Exons) undden Regulationssequenzen,die für die Transkriptiongebraucht werden. Kurz vordem Anfang des Insulingens(in der flankierenden Regionam 5’-Ende) liegen mehrereSequenzelemente, die fürdie Insulinproduktion ent-scheidend sind. VerschiedeneRegulationsproteine bindensich an diese Sequenzen undaktivieren sie. In Zellen, diekein Insulin herstellen, werdendiese DNA-Abschnitte durchandere Proteine blockiert.
Die RNA-Polymerase startetdirekt nach diesen Sequen-zen ihre Transkriptions-arbeit. Die Enden der tran-skribierten mRNA werdenbei Eukaryoten mit einer 5’-Cap („Kappe“) und einem 3’-Poly(A)-Schwanz modifiziert.Die Kappe soll vor Enzymab-bau (Phosphatasen undNucleasen) schützen und dieTranslation verstärken. DerPoly(A)-Schwanz ist nicht inder DNA codiert und stabili-siert offenbar die mRNA.
Auch nach der Translationam Ribosom kann dieGeschwindigkeit der Insulin-synthese noch gesteuertwerden; das Präproinsulin(128 Aminosäuren) ist längerals das aktive Hormon.Besondere Enzyme spaltenam Aminoende ein kurzesStück (24 Aminosäuren) ab,das als Signalsequenz für dieMembranpassage am endo-plasmatischen Reticulumdient. Anschließend wird ausdem so entstandenen Proin-sulin der mittlere Teil derPeptidkette (C-Peptid) ent-fernt. Die beiden kurzen A-und B-Ketten bilden das fer-tige Insulin. Sie werdendurch Disulfidbrücken zwi-schen den Cysteinen zusam-mengehalten.
Die Menge an aktivem Pro-tein wird häufig erst nachseiner Synthese durch Rück-kopplung im Körper regu-liert.
SH SH
S
S
SH
SH
Exon
A-Kette(21)
B-Kette(30)
spezifischer Enhancerder Inselzellen
Aktivatorproteine
ExonIntronRNA-
Polymerase
Transkription
Spleißen
Translation
Faltung des Proteins,Entfernung des Endabschnitts
NH2
COOH
COOH
Mittelabschnitt (C-Kette)35 Aminosäuren
mRNA, Cap am 5’ -Ende AA...
AA...reife mRNA
NH2
COOH
NH2 COOH
Präproinsulin-Polypeptid(110 Aminosäuren)
yy
S S
✂✂ ✂
COOHSH
Entfernungdes Mittelabschnitts
durch proteolytische Spaltung
Proinsulin-Polypeptid(86 Aminosäuren)
aktives Insulinprotein(51 Aminosäuren)
24 Aminosäuren dienen alsSignal für das Ausschleusen
Insulingen
Pankreas
Bauchspeicheldrüse
Langerhans-Insel(Insulin produzierende Zelle)
SS
✂
✂
NH2
NH2
NH2
COOH
SS
S
SS
Intron
C-Kette
S
(35 Aminosäuren)
SH
Bindung vonDisulfidbrücken
Abb. 3.37 Wie Proinsulin bildende Bakterienkolonienmithilfe von Antikörpernim Radioimmunoassay gefunden werden
ganen, sondern aus Bakterien stammte. Damit wur-de die erste, mithilfe der Gentechnologie hergestell-te Substanz am Menschen getestet. Zwei Jahre spä-ter erfolgte die offizielle Genehmigung für die medi-zinische Anwendung gentechnisch produziertenInsulins.
Der Bedarf an Insulin ist unglaublich hoch. Ein Dia-betiker brauchte zur Deckung seines Jahresbedarfsdie Bauchspeicheldrüsen von etwa 50 Schweinen.Die deutsche Firma Hoechst verarbeitete täglich elfTonnen Schweinebauchspeicheldrüsen, die vonmehr als 100 000 Schlachttieren stammten.
Ab 2005 wird in Deutschland kein tierisches Insu-lin mehr vertrieben.
� 3.15 Wie Insulin im Menschen
synthetisiert wird:
vom Präproinsulin über Proinsulin
zum aktiven Insulin
Insulin ist ein kleines Hormon, das aus zwei Pro-teinketten besteht, von denen die eine 21 (A-Kette)und die andere 30 Aminosäuren (B-Kette) lang ist.Von 1945 an hatte Fred Sanger (Box 3.4) in zehnJahre langer zäher Arbeit im Keller des Biochemi-schen Instituts in Cambridge (England) die Primär-struktur des Insulins erforscht.
Das Vorhaben der Insulinanalyse wurde seinerzeitals tollkühn angesehen. Kristallisiertes Rinderinsu-lin aus 120 Rindern dienten Sanger als Rohstoff.Fred Sanger bekam den Nobelpreis 1957, nur dreiJahre nachdem er die Sequenz aller 51 Aminosäu-rebausteine in den zwei Insulinketten entschlüsselthatte.
Beide Ketten werden zunächst als Bestandteileeiner längeren Kette von 110 Aminosäuren in denB-Zellen (β-Zellen) der Langerhans’schen Inseln imPankreas synthetisiert. Diese Langform ist das Prä-proinsulin (Abb. 3.36). Wie für andere Peptidhor-mone wird dieses Vorstufenprotein ins endoplasma-tische Reticulum sezerniert. Seine ersten 24 Ami-nosäuren dienen als Signal für die Zellmembranzum Ausschleusen des Insulins aus der Zelle. BeimDurchgang durch die Membran werden diese 24Aminosäuren durch Enzyme (Peptidasen) abge-trennt und verbleiben in der Zelle. Die restlichen86 Aminosäuren bezeichnet man als Proinsulin:B-Kette, C-Peptid plus A-Kette.
Anfangs- und Endstück dieses Moleküls (B und A)treten miteinander in Wechselwirkung und wer-den so durch zwei Disulfidbrückenbindungen(-S-S-) miteinander verknüpft. Danach wird derzentrale Teil des Proinsulins (C-Kette oder C-Pep-tid, mit 35 Aminosäuren) durch membranständigeEnzyme (Proteasen) im Golgi-Apparat der Zelleabgetrennt.
Die Bedeutung der C-Kette besteht darin, die A-und B-Ketten korrekt zueinander auszurichten.Ohne diese korrekte räumliche Faltung ist dasInsulin nicht voll aktiv.
Aktives Insulin und C-Peptid werden in Vesikelnaufbewahrt und nach Stimulus zusammen entlas-sen. Der „Tagesverbrauch“ von Insulin beträgtbeim Menschen etwa 1,8 mg.
Biotechnologie
68
Antikörper gegen ProinsulinKunststoffscheibe
Bakterienkolonie Proinsulin (Antigen)
Bindung von Proinsulin mit Fänger-Antikörper
Bindung der radioaktivenDetektor-Antikörper undanschließendes Auflegender Kunststoffscheibeauf Röntgenfilm
Lösung mit radioaktiv markierten Detektor-Antikörpern gegen Proinsulin
Schwärzung der Filmschichtdurch radioaktive Antikörper
Identifizierung der Proinsulin bildenden Kolonie
Sandwich
Abb. 3.38 Rosalyn Yalow(geb. 1921) entwickelte denRadioimmunoassay (RIA),Nobelpreis 1977
� 3.16 Der gentechnische Start
mit Ratten-Proinsulin
Um Insulin gentechnisch mithilfe von Bakterienzel-len zu produzieren, gingen Walter Gilbert (Abb.3.39) und Lydia Villa-Komaroff von der Harvard-Universität 1977 von einem Tumor der β-Zelleneiner Rattenbauchspeicheldrüse aus, also von tieri-schen Krebszellen. Als sie mit den Untersuchungenbegannen, waren Experimente mit menschlichenGenen noch nicht erlaubt.
Die mRNA der Krebszellen schrieben die Forschermit reverser Transkriptase in cDNA um, schnittendiese dann mit Restriktionsendonucleasen, um
klebrige Enden zu bekommen, und setzten dieDNA-Stücke in bakterielle Plasmide ein, die Penicil-lin- und gleichzeitig Tetracyclinresistenz-Gene ent-hielten (Abb. 3.34). Die mit der fremden DNA bela-denen Plasmide wurden dann in Bakterienzelleneingeschleust. Aus jeder dieser Zellen wurde einKlon gezüchtet. Man plattierte sie auf Agar-Nährbö-den aus, die Penicillin und Tetracyclin enthielten.
Würde einer dieser oben erwähnten Klone tatsäch-lich Ratten-Proinsulin produzieren? Um das zu prü-fen, beluden Gilbert und seine Mitarbeiter Kunst-stoffplatten mit Antikörpern gegen Proinsulin.Gegen Proinsulin lassen sich in Mäusen Antikörpererzeugen, wenn man diesen das Protein injiziert.
Die Wunder der Gentechnik
69
Box 3.3 Biotech-Historie:Insulinherstellung
1921 gelang den Kanadiern Frederick G.Banting (1891-1941, bei Flugzeugabsturzumgekommen) und Charles H. Best(1899-1978) in Toronto die Isolierung desInsulins aus tierischen Bauchspeicheldrüsen.Ihre Arbeit wurde als so epochal angesehen,dass Banting dafür nur zwei Jahre später,zusammen mit J. J. R. Macleod, den Nobel-preis für Physiologie oder Medizin erhielt.
Bereits 1922 wurde das Hormon an einemPatienten mit Erfolg klinisch erprobt. DieUS-Firma Eli Lilly & Co. (Indianapolis) bekamzunächst eine Exklusivlizenz für ein Jahrund produzierte „Iletin“ im Großmaßstab.
Das war der Startschuss für den heute nochzu den größten Insulinproduzenten der Weltgehörenden Konzern. Nach diesem Jahrwandte sich das Komitee der UniversitätToronto an verschiedene Interessenten. In Deutschland war das der Arzt Oscar Minkowski.
Minkowski fragte 1923 seinerseits bei denFarbwerken Hoechst an. Bei Hoechst hatteman schon zuvor aus Schlachthöfen inFrankfurt und Karlsruhe Drüsenmaterialgesammelt und versucht, Insulin zu gewin-nen. Anfang November 1923 kam dann dasselbstentwickelte „Altinsulin“ mit Genehmi-
gung aus Kanada auf den Markt. Fortan warHoechst führend in der Insulinforschungund bestimmt heute unter dem neuenNamen Sanofi-Aventis mit Eli Lilly, NovoNordisk und Berlin Chemie den Markt inDeutschland.
Viele andere Firmen versuchten ihr Glück,scheiterten aber zumeist am Rohstoffpro-blem: Das Einsammeln des Materials ausvielen kleinen deutschen Schlachthäusernwar mühsam – man denke dagegen an dieriesigen von Upton Sinclair beschriebenenSchlachthöfe von Chicago! Bauchspeichel-drüsen mussten importiert werden.
1936 gelang es dann der Firma Hoechst alserster, die gesamte Produktion auf das kris-tallisierte Hormon umzustellen. Die daraushergestellten Lösungen waren besser vonbegleitendem Fremdprotein gereinigt undsomit verträglicher. Auch an löslichenDepot-Insulinen wurde gearbeitet. 1938kam das „Depot-Insulin Hoechst“ mit demStabilisator Surfen auf den Markt. Sogarwährend des Krieges blieb die Versorgungdurch ein neues Verfahren zur Drüsenkon-servierung gesichert. In den ersten Jahrennach 1945 erreichte die Produktion jedoch
einen Tiefstand. Hoechst blieb allerdingsHauptlieferant und brachte 1953 das lang-wirkende „Long-Insulin“ auf den Markt.Dann gelang nach 10-jähriger Arbeit Frede-rick Sanger die Bestimmung der Insulin-struktur (siehe Box 3.4).
1963-1965 wurde in verschiedenen Arbeits-gruppen die Insulin-Totalsynthese ausge-führt, und 1969 benutzte DorothyCrowfoot-Hodgkin die Röntgenstruktur-analyse zur Aufklärung der Raumstruktur.
Frederick Banting undCharles Best
Oscar Minkowski(1858-1931) fand1889 an der Medizi-nischen Klinik inStraßburg, dassHunde Diabetesentwickeln, wennihnen das Pankreasentfernt wird. Erüberzeugte Hoechst1923 zur Insulin-produktion.
0ben: Insulin-produktion beiHoechst links: Werbungfür Insulin vonHoechst
Abb. 3.39 Walter Gilbert(geb. 1932), Nobelpreis fürChemie 1980
7.1 Mikroben sind essbar! 172
7.2 Algen und Cyanobakterien 172
7.3 Single cell-Protein: Hoffnung
auf billige Eiweißquellen 174
7.4 Mycoprotein ist als pflanz-
liches Eiweiß beim Verbraucher
erfolgreich 175
7.5 „Grüne“ Biotechnologie
ante portas! 178
7.6 Felder im Reagenzglas:
in vitro-Pflanzenzucht 178
7.7 Meristemkultur 179
7.8 Haploidenkulturen: Staub-
beutel und Fruchtknoten 180
7.9 Kallus- und Suspensions-
kulturen 181
7.10 Pflanzenzellen im Bioreaktor
produzieren Wirkstoffe 183
7.11 Welche Pflanzenwirkstoffe
werden dem Shikonin
folgen? 184
7.12 Agrobacterium – ein Schädling
als Gentechniker 185
7.13 Biolistischer Gentransfer: DNA-
Schuss aus dem Revolver 185
7.14 Transgene Pflanzen: Herbizid-
resistenz 188
7.15 Biologische Insektentöter 189
7.16 Blaue Nelken und Anti-Matsch-
Tomaten 193
7.17 Gefahr durch Gen-Food? 194
7.18 Soll man Gen-Food
kennzeichnen? 195
7.19 Gene-Pharming 195
7.20 Transgene Pflanzen – eine
hitzige Debatte 198
7.21 Tropische Palmen in
Deutschland? 198
7.22 Bakterien in Schneekanonen
sichern den Skiurlaub 200
Kapitel 7
Grüne Biotechnologie
� 7.1 Mikroben sind essbar!
Für die Produktion von einem Kilogramm tieri-schem Protein werden fünf bis zehn KilogrammPflanzeneiweiß benötigt. Hierbei geht Eiweiß mas-siv verloren, zusätzlich zu den riesigen Verlustendurch Schädlinge, Ernte, Transport und Lagerung.Mikroorganismen könnten wirksam helfen: Sieproduzieren nicht nur Medikamente, Wein undKäse – Mikroben selbst sind essbar! Sie enthaltenwertvolle Proteine, Fette, Zucker und Vitamine.
Schon 1521 beschrieb der Spanier Bernal Diaz delCastillo nach der Eroberung Mexikos, dass dieAzteken merkwürdige kleine käseähnliche Kuchenaßen. Diese Kuchen bestanden aus in mexikani-schen Seen wachsenden mikroskopisch kleinenAlgen, von den Azteken Techuilatl genannt. Eshandelte sich dabei um Spirulina. Spirulina ist kei-ne „echte“ Alge, vielmehr ein Cyanobakterium,eine „Blaubakterie“.
Im Kaiserreich Montezumas sollen Diener, die denHerrscher täglich mit frischem Fisch versorgenmussten, was nur im Dauerlauf über sehr großeEntfernungen möglich war, als Kraftnahrung Spiru-lina verwendet haben. Noch zu Cortez’ Zeitenwurde es auf den Märkten der Einheimischengehandelt und als Beigabe zu Brot und Körnerspei-sen gegessen. Spirulina schöpften die Fischer mitfeinmaschigen Netzen aus Salzseen ab (Abb. 7.1),die damals noch nicht trocken gelegt waren. Heutegibt es Spirulina wohl nur noch im Texcoco See inMexiko. Die anderen Seen, mitsamt den schwim-menden Gärten der Azteken, sind heute unfrucht-bare Wüste.
Tausende Kilometer entfernt verzehren die Einge-borenen am Tschadsee in Afrika (Nigeria) ebenfallsseit Urzeiten Spirulina. Auf lokalen Märkten amTschadsee gibt es dünne, harte, blau-grüne Algen-kuchen zu kaufen. Das Volk der Kanembu nenntdieses Produkt Dihé. Dihé ist wichtiger Bestandteilin 70% aller Gerichte. Es wird als Beimischung zuSoßen aus Tomaten, Chillis und diversen Gewür-zen verarbeitet, die zusammen mit dem Grundnah-rungsmittel Hirse gegessen werden. Die Spirulina-Kuchen stellt man durch Sonnentrocknung her.Zuvor werden die schwimmenden Algen ausgeschützten Bereichen des Sees aus dem Wassergeschöpft. Dann lässt man sie abtropfen und breitetsie im warmen Sand aus, wo sie rasch trocknen.
In westlichen Ländern und Japan isst man Spiruli-na aus Algenfarmen als Cholesterin senkende, blut-reinigende Diät (Abb. 7.4). 100 Gramm Spirulinasollen rund 70 Gramm Protein, 20 Gramm Zucker,zwei Gramm Fasern und nur zwei Gramm Fett ent-
halten, aber wichtige Vitamine (A, B1, B2, B6, B12, E)und Mineralstoffe.
� 7.2 Algen und Cyanobakterien
Algen sind – mit Ausnahme der prokaryotischenSpirulina – photosynthetische Eukaryoten (Box7.1). Makroalgen sind ökonomisch bedeutsamerals Mikroalgen: Grüne (Chlorophyta), Rote (Rho-dophyta) und Braune Makroalgen (Fucophyta oderPhaeophyta) werden gegenwärtig genutzt.
Braunalgen, wie den bei Tauchern beliebtendschungelartig wachsenden kalifornischen Kelp(Macrocystis) (Abb. 7.2), erntet man seit 1900. Ausihm wurde seit 1921 in San Diego das gelatinearti-ge Alginat produziert. Hier gibt es Riesentangwäl-der. Heute wird Alginat als Eindicker und Stabilisa-tor in Nahrungsmitteln und Eiscreme, in der Textil-industrie und als Verkapselung für Medikamente(und für Enzyme und Hefen, Kap. 2) genutzt.
Andere Algenprodukte sind das Gel bildende Agar(früher Agar-Agar genannt, wichtig für die Kultivie-rung von Mikroben und die Gelelektrophorese, sie-he Kap. 10) und Carrageenan.
Die geschmacksverstärkende Aminosäure l-Gluta-mat wurde zuerst in Algen in Japan gefunden (Kap.4). Andere Braunalgen wie Undaria (jap. wakame)und Laminaria (jap. konbu) wachsen an den Küs-ten Japans und Chinas und werden für Salate, Sup-pen, Nudeln oder mit Fleisch genutzt (Abb. 7.5 und7.6). Der jährliche Marktwert beider Algen liegt bei600 Millionen US-Dollar. 20 000 Tonnen Wakamewerden jährlich geerntet.
Die Rotalge Porphyra (jap. nori) wird in Japan seitdem Mittelalter kultiviert. Heute wird sie in riesi-gen Mengen an Bambusbüscheln oder horizonta-len Netzen in Meeresfarmen gezogen und späterluftgetrocknet.
Die wichtigsten Mikroalgen stammen aus zweiverschiedenen Klassen: Die schon erwähnten pro-karyotischen Blaubakterien und die eukaryotischenGrünalgen.
Zu den früher als Blaualgen bezeichneten prokaryo-tischen Blaubakterien (Cyanobakterien) gehörendie wirtschaftlich wichtigen Arten der Gattung Spi-rulina. Der in Asien bei den Reisbauern kultivierteWasserfarn Azolla beherbergt die symbiotischeBlaubakterie Anabaena azollae (Abb. 7.9), auchSchnurfaden genannt, die den Farn mit Stickstoffversorgt.
Die Spirulina-Zellwand besteht wie die der Bakte-rien aus Mucoproteiden und ist daher durchmenschliche Verdauungsenzyme leicht aufzu-
Biotechnologie
172
Abb.7.4 Spirulina-Tabletten
Abb. 7.1 Azteken beim Abfischen von Spirulina
Abb. 7.3 Spirulina-Farm inIndien
Abb. 7.2 Braunalge Kelp(Macrocystis)
Grüne Biotechnologie
173
Box 7.1 Photosynthese
Fast alle freie Enthalpie, die von biologischenSystemen verbraucht wird, stammt von derEnergie der Sonne. Das sind riesige Mengen:etwa 4 ·1017 kJoule oder 1010 Tonnen proJahr in Zucker umgewandelter Kohlenstoff.Die Sonnenenergie wird durch die Photosyn-these in chemische Energie umgewandelt.Wasser und Kohlendioxid vereinigen sich ineinem hochkomplexen Prozess zu Kohlen-hydraten (zunächst Glucose, dann Saccharo-se und Stärke) und molekularem Sauerstoff.
In den Chloroplasten der grünen Pflanzenerzeugen Pigmentmoleküle (Chlorophylle)in der Thylakoidmembran aus eingefangenerLichtenergie Elektronen hoher Energie. Sie werden in der Lichtreaktion zur Erzeu-gung von NADPH+H+ und ATP (Kap. 1) verwendet.
Die Photosynthese der grünen Pflanzen wirddurch zwei miteinander verbundene Photo-systeme ausgeführt. Sehr stark vereinfachtstellt man sich das so vor:
Im Photosystem II führt die Lichtanregungvon P680 (einem Paar von Chlorophyll-Molekülen) zu einem Elektronentransferüber mehrere pigmentierte Moleküle aufPlastochinon A und dann auf Plastochinon B.Diese energiereichen Elektronen werdendurch Entzug von Elektronen niedrigerEnergie aus Wassermolekülen wiederersetzt: Das Sauerstoff entwickelnde Zen-trum entnimmt Wasser ein Elektron, transfe-riert es zu einer Tyrosingruppe und diesebringt es zurück zum Chlorophyll, dasdadurch ein weiteres Photon aufnehmenkann.
Für jeweils vier übertragene Elektronen wirdein Molekül Sauerstoff erzeugt. Vom Plasto-chinon laufen die Elektronen über einenCytochrom-bf-Komplex zum Plastocyaninund von dort zum Photosystem I.
Das hier gezeigte Photosystem I ist ein tri-merer Komplex, der in der Membran„schwimmt“. Jeder der drei Untereinheitenhat hunderte Cofaktoren (grün Chlorophyll,orange Carotinoide). Die Farben haben eineBedeutung: Chlorophyll absorbiert blauesund rotes Licht – deshalb sehen wir Pflan-zen in wunderbarem Grün.
Das Photosystem I besitzt Elektronentrans-ferketten als Zentrum der drei Untereinhei-ten. Jede ist von einem dichten Ring ausChlorophyll- und Carotinoid-Molekülenumgeben, die als „Antennen” fungieren.
Diese Antennen absorbieren Licht und trans-ferieren Energie an ihre Nachbarn. Dannwird alles in die drei Reaktionszentren kana-lisiert, die Elektronen (eine reduzierendeKraft) generieren.
Summa summarum führt die Lichtreaktionin den Thylakoidmembranen der Chloro-plasten erstens zu einer reduzierenden Kraft(zur Erzeugung von NADPH+H+), zweitenszur Bildung eines Protonen- (H+)Gradienten(ein Gefälle zwischen den beiden Seiten derThylakoidmembran, damit wird ATPerzeugt) und drittens zur Produktion vonSauerstoff.
Eine weitere Stufe ist die CO2-Fixierung:Das Enzym Ribulose-1,5-bisphosphat-Carb-oxylase/Oxygenase (kurz RUBISCO) ist eine
Lyase (Kap. 2) und bildet die Brücke zwischenLeben und Leblosem.
Es verbindet das anorganische CO2 mitRibulose-1,5-bisphosphat, einer kurzenZuckerkette mit fünf Kohlenstoffatomen.RUBISCO bildet daraus zwei 3-Phosphogly-cerate (mit je drei Kohlenstofftomen). Diemeisten Phosphoglycerat-Moleküle werdenrecyclisiert, um noch mehr Ribulose-bisphosphat zu bilden, aber jedes sechsteMolekül wird zur Bildung von Saccharoseoder Stärke (als Speicherstoff) verwendet.
16% der Eiweiße der Chloroplasten sindRUBISCO und bei der gewaltigen Menge anPflanzen ist RUBISCO offenbar das häufigsteProtein auf der Erde!
Ribulose-1,5bis-phosphat
H2O
CO2
P
P
RUBISCO
3-Phosphoglycerat
P
2 NADPH+2H+
2 ATP
ATP
Stärke,Zucker
P
Licht
Licht
Licht
photo-synthetisches
Reaktions-zentrum
Licht sammelnder
Komplex
PS I
PS IILicht
Oben rechts: Struktur desPhotosystems I und II Links: Prozess der CO2-Fixierung durch RUBISCO
Abb. 7.5 GetrockneteMakroalgen dienen in Chinaund Japan als preiswerteGrundlage für Suppen.
schließen, ein Vorteil bei ihrer Verwendung fürDiätzwecke. Spirulina ist ein fädiger, schraubiggewundener Organismus, der aus 150 bis 300 Ein-zelzellen bestehen kann und dann eine Länge vonbis zu einem halben Millimeter erreicht. Zu denBlaubakterien gehört auch der Zittertang oderSchleimling Nostoc (Abb. 7.9).
Zu den eukaryotischen Grünalgen (Chlorophy-ceae) zählen die Arten aus den Gattungen Chlorel-la, der Vierling Scenedesmus, die begeißeltenDunaliella und Chlamydomonas (Abb. 7.9) und dieaus bis zu 20 000 Zellen bestehende AlgenkolonieVolvox. Volvox ist hoch organisiert, mit differen-zierten Zellen. Sie sind wichtige Algen im Abwas-ser und Plankton der Seen und Meere.
Die Zellen von Chlorella und Scenedesmus sinddurch eine feste Cellulosewand mit Sporopollenin-Einlagerungen (der Substanz der Wände von Pol-lenkörnern höherer Pflanzen) umhüllt. Die damitimprägnierten Zellwände sind chemisch besondersschwer angreifbar. Deshalb müssen die etwa zehnMikrometer großen Zellen vor der Weiterverwen-dung zu Ernährungszwecken aufgebrochen wer-den.
Algen wie Chlorella werden heute mit Kosten vonrund zehn Euro je Kilogramm gezüchtet und vorallem als Diätnahrung zum etwa zehnfach höherenPreis verkauft. Der Rohproteinanteil dieser Algenbeträgt etwa 50% der Gesamtmasse (im Vergleichdazu bei Sojabohnen 35%), und sie weisen einenhohen Anteil an ungesättigten Fettsäuren und Vita-minen auf.
Dunaliella -Arten, einzellige, bewegliche Flagella-ten, besitzen hingegen keine feste Zellwand. Ihreauffälligste Eigenschaft ist eine außergewöhnlicheSalztoleranz. Diese halophilen Algen treten dahermassenhaft in eintrocknenden Lagunen von Meer-wassersalinen auf. Wegen ihres enormen Gehaltsan β-Carotin färben sie die Salzlauge tiefrot (Abb.7.7). Zum Ausgleich des externen osmotischenWertes reichern sie in beträchtlichem Maße Glyce-rin an, das wie das Carotin industriell gewonnenwird.
Algen verdoppeln ihre Masse in nur sechs Stunden.Gräser brauchen dafür zwei Wochen, Kücken vierWochen, Ferkel sechs Wochen und Kälber zweiMonate.
In vielen Ländern wird deshalb an Algenfarmengearbeitet. Man braucht dazu große, flache Wasser-becken, in denen die Algen genügend Sonnenlichtbekommen (Abb. 7.3 und 7.7). Mit dessen Hilfe bil-den sie aus Kohlendioxid, Wasser und Nährsalzen
Zucker und aus den Zuckern dann Eiweiß. Lichtund Luft sind kostenlos, und nur wenige billigeNährsalze werden benötigt, um die Algen üppigwachsen zu lassen. Je Hektar Fläche ergibt Spiruli-na etwa zehnmal mehr Biomasse als Weizen undhat einen viel höheren Eiweißgehalt als dieser. Beider Ernte werden die Algen herausgesiebt, an derLuft getrocknet und dann, nachdem sie mitGeschmacksstoffen versetzt wurden, verkauft.
Warum gibt es dann noch keine riesigen Algenfar-men in den Hungergebieten? Hier fehlt selbst dieeinfachste Technik dazu; in vielen Gebieten istzudem das Wasser knapp und teuer. Der entschei-dende Faktor: Unter günstigen Produktionsbedin-gungen kostet Algenprotein zehn US-Dollar proKilogramm und Sojaeiweiß nur 20 Cent pro Kilo-gramm. Fachleute sind dennoch überzeugt, dassdie Algen eine große Zukunft haben werden.
Noch schneller als Algen wachsen Bakterien, Hefenund Pilze. Bakterien verdoppeln ihre Masse im Zeit-raum von 20 Minuten bis zwei Stunden, und siekönnen zu 70% aus Eiweiß bestehen. Im Durch-schnitt können Hefen je Masseeinheit 100 000-malschneller Protein bilden als eine Kuh. Dabei gibteine Kuh nur ein Elftel der aufgenommenen Nähr-stoffe aus der Pflanzennahrung in Form von Fleischan uns weiter. Zehn Elftel gehen also beim Rind fürdie menschliche Ernährung verloren. Das ist beiden Bakterien, Hefen und Pilzen anders: Hier wirdfast die gesamte Nährstoffmenge in für Menschenund Tiere verwertbare Eiweiße, Zucker und Fetteumgewandelt. Es lag nahe, das zu nutzen.
� 7.3 Single cell-Protein: Hoffnung
auf billige Eiweißquellen
Die moderne Geschichte der Eiweißproduktiondurch Mikroben begann im kaiserlichen Deutsch-land während des Ersten Weltkriegs mit der Zuchtvon Hefen. Wegen der Lebensmittelknappheitzüchtete man Bäckerhefe im Großmaßstab und„streckte“ damit hauptsächlich Wurst und Suppen.Hefen haben den großen Vorteil, dass sie sich vonbilligen, sonst nicht verwertbaren zuckerhaltigenLösungen ernähren und den Zucker direkt in wert-volles Protein umwandeln. Im 900 Tage belagertenLeningrad bewahrten Hefen Tausende von Men-schen im Zweiten Weltkrieg vor dem Hungertod.Kurz nach dem Krieg stillten in Deutschland „Hefe-flocken“ den Hunger vieler Menschen.
Erst in den 60er-Jahren begann man in Europaerneut, Anlagen zur Eiweißproduktion durchMikroben zu errichten (Box 7.2). Der Proteinbe-darf der Menschheit stieg. Man rechnete mit Hun-
Biotechnologie
174
Abb. 7.8 Fusarium gramina-
reum, der Erzeuger vonQuorn
Abb. 7.6 In Japan gehörenMakroalgen seit jeher zumfesten Bestand der Ernäh-rung.
Abb. 7.7 Die Alge Dunaliella
wird in Farmen kultiviert(oben: in Westaustralien); unten: Carotinoide reichernsich in der rechts gezeigtenZelle an.
Abb. 7.9 Algen und Cyanobakterien
gersnöten in der Zukunft und hatte inzwischen ent-deckt, dass sich Mikroorganismen nicht nur vonzuckerhaltigen Nährlösungen, sondern auch vonkohlenwasserstoffhaltigen Bestandteilen des Erd-öls, Alkanen (Paraffinen), und von Methanol ernäh-ren können. Die wachsartigen Alkane sind für Men-schen und Tiere nicht verwertbar, nur Mikrobenkönnen sie in wertvolles Eiweiß umwandeln.
Im Osten Europas konzentrierte man sich in derHoffnung auf permanent billiges Erdöl auf Alkanhe-fen (Candida), im Westen, vor allem die britischeICI, auf Methanol verwertende Hefen und Bakte-rien (siehe Box 7.2 und Abb. 7.10). Die beiden sohoffnungsvollen Riesenprojekte endeten letztlichohne Erfolg.
Die Alkanhefen wurden nur begrenzt als Futtermit-tel freigegeben, denn man befürchtete Krebs erre-gende Rückstände. Beide Projekte in Ost und Westscheiterten letztlich ökonomisch an den zwei Erd-ölkrisen. Im Westen scheiterte das Methanol-Fut-
termittel noch zusätzlich an EU-Subventionen.Denn diese Subventionen machten Magermilch-pulver als Futtermittelzusatz unerreichbar preis-wert.
Die Biotechnologen sammelten jedoch unschätzba-re Erfahrungen beim Bau und Betrieb von riesigenBioreaktoren.
� 7.4 Mycoprotein ist als pflanzliches
Eiweiß beim Verbraucher
erfolgreich
Ein sehr erfolgreiches Produkt ist dagegen dasMycoprotein (griech. mykes, Pilz) von Rank HovisMcDougall (RHM) und heute Marlowe Foods, eineTochter der ICI. RHM, der viertgrößte Nahrungs-mittelproduzent Westeuropas, fand seine Mikrobein den 60er-Jahren und wendete insgesamt über 30Millionen Pfund Sterling für den Pilz auf, der in pas-sable Imitationen von Fisch, Geflügel und Fleischverwandelt werden kann (Abb. 7.13).
Grüne Biotechnologie
175
Anabaena
Chlorella
Nostoc
Volvox
Dunaliella
Spirulina
Scenedesmus
Spirogyra
Chlamydomonas
Abb. 7.10 Das BakteriumMethylophilus methylotro-
phus produziert hochwerti-ges Protein aus Methanol.
Biotechnologie
176
Box 7.2 Biotech-Historie:
Einzellerprotein
In der Sowjetunion startete man in den60er-Jahren unter Nikita Chrustschow,beflügelt einerseits von Weltraumerfolgenund andererseits geplagt von ständigen Miss-ernten, ein aufwändiges Programm zurSuche nach den besten Alkanenfressern. DieIdee war, aus billigem Erdöl wertvollesEiweiß gewinnen.
Man suchte auch parallel nach Möglichkei-ten, die Cellulose sibirischer Wälder zuZucker abzubauen und diesen dann zu „ver-hefen“ und Eiweiß zu gewinnen. Schon1963 begannen erste Versuchsanlagen zuarbeiten. Auf vorgereinigten Erdölprobenwuchsen Hefestämme der Gattung Candida,die „mit Heißhunger“ Alkane verzehrten.Bei Beginn der Hefeproduktion aus denAlkanen des Erdöls gab es Bedenken vonÄrzten und Tierärzten, die meinten, wegender schweren Verdaulichkeit von Alkanenfür Menschen und Tiere könnte das Alkan-hefeeiweiß für höhere Lebewesen problema-tisch oder sogar Krebs erregend sein. Lang-jährige russische Experimente zeigten zwar,dass das Hefeeiweiß wohl unbedenklich indie Nahrungskette für den Menschen aufge-nommen werden kann. Im Westen stieß das(wahrscheinlich zu Recht) jedoch auf großeSkepsis. Das erste große Werk für Alkanhefebegann in der Sowjetunion 1973 mit derProduktion von 70 000 Tonnen Hefe imJahr. Die Anlage im Petrolchemischen Kom-binat Schwedt, in der damaligen DDR, demEndpunkt der sowjetischen ErdölleitungDrushba (Freundschaft), begann Anfang1986 ihren ständigen Betrieb. Sie liefertejährlich mit Tauchstrahlreaktoren 40 000Tonnen des Futterhefepräparates Fermosin®.Die Bioreaktoren waren zweifellos eineMeisterleistung der ostdeutschen Ingenieureund Biotechnologen. Nach der deutschenWiedervereinigung wurde der Fermosin®-Prozess gestoppt.
Aber auch im Westen scheiterten single cellProtein-Projekte: British Petroleum (BP)
beteiligte sich 1971 auf Sardinien an derHerstellung von Toprina®, einem Erzeugnisaus Hefe, die auf Resten von Rohöl wuchs,durch die italienische Firma ANIC. AlsSchuldige für das Scheitern des Projektswurden ausgemacht: Die Erdölkrise, dieSoja-Lobby, die Sojapreise reduzierte, dieDiskussion über die Unbedenklichkeit vonToprina® (hoher Gehalt an Nucleinsäuren,ruft Gicht hervor) und Umweltbedenken.
Parallel wurde im Westen Europas an derVerwertung von Methanol geforscht. Aufeinem Rugbyplatz in der britischen Graf-schaft Durham wurden die Biotechnologender britischen Imperial Chemical Industries(ICI) fündig. Sie entdeckten das BakteriumMethylophilus methylotrophus (Abb. 7.10).
Etwa 10 000 Mikroorganismen waren 13Jahre lang gesucht und getestet worden aufder Suche nach einem Mikroorganismus,der schnell auf petrolchemischen Rohstoffenwächst und ein Eiweißkonzentrat für Haus-tiere liefert: Pruteen® war das Resultat.Zuerst hatte sich ICI auf Methan als Kohlen-stoffquelle konzentriert, weil die FirmaZugang zum reichlichen Nordseegas hatte.Das schien ein eleganter Weg zu sein vomeinfachsten organischen Molekül zum kom-plizierten Protein. Nicht nur die Explosions-gefahr des Methans, sondern auch seinegeringe Löslichkeit und das Problem, es imMedium gleichmäßig zu verteilen, sprachenaber gegen Methan. Methanol, oxidiertes(also Sauerstoff enthaltendes) Methan, kanndagegen den Sauerstoffbedarf der Mikrobenleichter stillen, ist unbegrenzt wasserlöslichund führt nicht zu einer so hohen Wär-meentwicklung im Bioreaktor.
Die ICI-Forscher entschieden sich auch des-halb für den Methylophilus -Stamm (lat.Methanol liebend), weil er stabil und freivon toxischen Nebeneffekten war. Die Ent-scheidung, eine Reinkultur der Bakterien ineinem kontinuierlichen Prozess zu züchten,hatte allerdings eine Konsequenz: Der Pro-zess musste unter den außergewöhnlichen
Bedingungen der Sterilität geführt werden!Im Gegensatz dazu lief der Alkanhefeprozessunsteril, das heißt, der Hefestamm verdrängtselbst alle Konkurrenten – so wie das beiden meisten biotechnologischen Prozessenzur Nahrungsmittelproduktion der Fall ist.
Die britische Firma John Brown Engineersand Constructors baute das gigantischeWerk von ICI bei Billingham für den größtenSteril-Bioprozess der Welt. Die Biofabrikbedeckte eine Fläche von acht Hektar. DerBioreaktor, ihr Kernstück, war 60 Meterhoch (mit acht Blasensäulen-Fermentern)und enthielt 150 000 Liter absolut keimfrei-er Nährlösung, in der die Methanolbakterienleben. Er wurde durch ein ausgeklügeltesSystem von 20 000 Ventilen und Filtern dreibis vier Monate lang ununterbrochen freivon fremden Mikroben gehalten. Methylo-philus lebt bei 35°C nur von Methanol,Ammoniak und Luftsauerstoff. Ständig wur-den dem Bioreaktor Mikroben entnommen,mit heißem Wasserdampf abgetötet, zu grö-ßeren Klumpen zusammengeballt undgetrocknet. Sie ergaben das körnige, kara-melfarbige Produkt Pruteen®. Alles schien inbester Ordnung.
Als das Werk 1976 startete, wurde die Firmajedoch mit den steigenden Energiepreisenund einer hervorragenden Sojaernte kon-frontiert; Einzellerprotein konnte so nichtökonomisch produziert werden. Methylo-philus wurde deshalb sowohl gentechnischals auch mit Methoden der klassischenGenetik verbessert: Das Gen für das EnzymGlutamat-Dehydrogenase (für eine effektive-re Stickstoffverwertung aus Ammoniak)wurde erfolgreich eingeschleust. Die Eiweiß-ausbeute konnte so um fünf bis sieben Pro-zent verbessert werden. Das ICI-Werk funk-tionierte, die Nachfrage blieb jedoch hinterden Erwartungen zurück. Die bis 1982investierten 100 Millionen Pfund Sterlingbetrachtet ICI dennoch als „Eintrittskartezur Biotechnologie“.
Wichtige Erfahrungen flossen aber in diegroßtechnische Realisierung der Pilzeiweiß-Quorn -Produktion (siehe Haupttext) ein.
Alkanhefe-Bioreaktor (42 m hoch) in Schwedtzu DDR-Zeiten
Das riesige ICI-Werk bei Billingham, natürlicheBioreaktoren im Vordergrund
Einer der ICI-Bioreaktoren
Forscher von RHM hatten mehr als 3 000 Boden-proben aus der ganzen Welt gesammelt. Wie so oftlag der Haupttreffer aber ganz in der Nähe: In derNachbarschaft des Ortes Marlowe in Buckingham-shire, England, wurde Fusarium graminareum(heute: Fusarium venenatum, Abb. 7.8) gefunden.Zuvor war sein Name nur Pflanzenpathologengeläufig gewesen: Der Pilz verursacht Wurzelfäulebei Weizen.
RHM produzierte damals 15% des britischen Spei-sepilzangebotes. Wegen schlechter Erfahrungenmit psychologischen Vorurteilen der Konsumentengegen Bakterieneiweiß betonte RHM von Anfangan: Fusarium sei ein Pilz wie unsere Speisepilzeund Trüffel, die wir essen, ohne zweimal nachzu-denken.
Abgesehen davon, dass Fusarium fast geruch- undgeschmacklos ist (Abb. 7.11), ideal für Fleischimita-te, enthält er auf Trockengewicht gerechnet etwa50% Protein, eine Zusammensetzung wie die vongegrilltem Beefsteak. Der Pilz hat aber einen nied-rigeren Fettgehalt als Beefsteak, nur 13%, noch dazupflanzliches Fett, kein Cholesterin (dafür Ergoste-rin) und einen Fasergehalt von 25% – das alles zähltzunehmend bei Gesundheitsbewussten (Abb. 7.12).
Ein Hauptvorteil bei der Gewinnung von Pilz-gegenüber Bakterienzellen ist, dass sie typischer-weise viel größere Dimensionen besitzen, also ausdem Fermentationsmedium leicht abtrennbar sind.Andererseits wachsen aber Pilze viel langsamer alsBakterien mit einer Verdopplungszeit von vier bissechs Stunden im Vergleich zu 20 Minuten bei Bak-terien.
Auch das kann sich in einen Vorteil verwandeln:Langsameres Wachstum bedeutet auch, dass imEndprodukt weniger Nucleinsäuren enthaltensind. Nucleinsäuren, während eines längeren Zeit-raums in hohen Konzentrationen durch Säugetiereund den Menschen aufgenommen, führen zuGicht.
Während einige Bakterien 25% Nucleinsäuren undHefen bis zu 15% enthalten, gelang es RHM bei derneuen Nahrung Mycoprotein, den Gehalt auf weni-ger als die für den Menschen akzeptable Grenzevon 1% zu senken. Der Pilz hat auch eine Amino-säurezusammensetzung, die von der UN-Welter-nährungsorganisation (FAO) als „ideal“ empfohlenwird.
Die vielleicht außergewöhnlichste Eigenschaft desPilzes ist der Weg, wie er in ein komplettes Imitat-Spektrum von Nahrungsmitteln, von Suppen undBiskuit bis zu überzeugenden Nachahmungen vonGeflügel, Schinken und Kalbfleisch, verwandeltwerden kann (Abb. 7.11 bis 7.13).
Der Schlüssel zu dieser Anpassungsfähigkeit: DieLänge der Fasern kann kontrolliert werden; je län-ger der Pilz im Bioreaktor wachsen „darf“, umsolänger sind auch die Fasern, und desto gröber ist dieTextur des Produktes. Das Medium besteht ausGlucosesirup als Kohlenstoffquelle mit Ammoniakals Stickstoffquelle. Der Sirup kann aus allen verfüg-baren Stärkeprodukten (Kartoffeln, Weizen,Maniok) gewonnen werden, und der Prozess istsehr viel effizienter als die Umwandlung von Stär-ke in Protein durch Haustiere.
Zur Pilzproduktion taten sich ICI und RHM zusam-men. 1985 wurde das Mycoprotein in Großbritan-nien vom MAFF (Ministry of Agriculture, Fisheriesand Food) freigegeben. Das erste Produkt war einSavory Pie. In den 90er-Jahren wurde MarloweFoods in Marlowe gegründet. Inzwischen ist dasPilzeiweiß in England als Quorn auf dem Markt.
Wurden vor 1993 noch Umsätze von weniger alsdrei Millionen US-Dollar pro Jahr erzielt, lagen sie2001 dank mächtiger Werbung bei 150 Millionen.Interessant sind die Zielgruppen: Frauen von 25 bis65 in den USA und von 25 bis 45 in Großbritan-nien. Die ältere Generation ist eben konservativ,obwohl die Firma gezielt ungenau nicht von Mikro-ben, sondern von „pflanzlichem Eiweiß“ spricht.
Grüne Biotechnologie
177
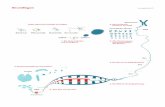
Abb. 7.14 Links: Wie Pflanzen in vitro vermehrtwerden könnenKarottenwurzel
Segmentierung
Fragmentewachsen in
Nährmedium
Teilung derfreien Zellen
embryoartigeEntwicklung
Agarmedium
reife Pflanze
✂
Abb. 7.13 Quorn-Produkteaus Fusarium: Fleischbäll-chen und Würstchen ohneFleisch
Abb. 7.12 Erstes Probeessenvon „veredeltem“ Mycopro-tein
Abb. 7.11 Mycoprotein vorder Verarbeitung. Links„Rind“, rechts „Huhn“































![Studie – BPM Suites 2013 [PDF 3.29 MB] - iese.fraunhofer.de · Studie – BPM SuiteS 2013 Fraunhofer IESE Dr. Sebastian Adam Norman Riegel Thomas Jeswein Matthias Koch SP Consulting](https://static.fdokument.com/doc/165x107/5afe607f7f8b9a994d8ee118/studie-bpm-suites-2013-pdf-329-mb-iese-bpm-suites-2013-fraunhofer.jpg)

